作者简介:席刚俊(1981—),男,湖北仙桃人,硕士,助理研究员,从事铁皮石斛、白芨病害研究。E-mail:2469250487@qq.com
为研究白芨内生菌的多样性,从新鲜白芨根中提取内生真菌的DNA,构建ITS rDNA文库,进行高通量测序和多项信息分析。结果显示,白芨内生真菌主要属于担子菌门(83.4%)和子囊菌门(14.3%),另外2.3%无法鉴定;担子菌门、子囊菌门、接合菌门的菌根优势度指数分别为0.834 1、0.142 6、0.000 3,无法鉴定的真菌菌根优势度指数为0.022 9。纲、目的分类水平上,伞菌纲蜡壳菌目占83.4%,盘菌纲盘菌目占11.4%,锤舌菌纲柔膜菌目占2.1%,无法鉴定的占2.3%。科分类水平上,内生真菌主要属于Serendipitacea(83.4%)和盘菌科(11.4%)。属的分类水平上,梨形孢属( Piriformospora)占83.4%。Shannon index、Chao1、Observed species和Simpson 4种曲线分析结果显示,白芨菌根内生真菌的多样性呈均匀分布。
To research the diversity of endophytic fungi in Bletilla striata, DNA of endophytic fungi was extracted from the fresh root of Bletilla striata, and ITS rDNA library was constructed. Then, this library was analyzed by high-throughput Illumina sequencing technology and bioinformatics. The results showed that endophytic fungi of Bletilla striata were mainly distributed in Basidiomycota (83.4%) and Ascomycota (14.3%), the other 2.3% could't be identified. Dominance index of endophytic fungi were Basidiomycota (0.834 1), Ascomycota (0.142 6), Zygomycota (0.000 3), and unidentified (0.022 9). At Class and Order levels, the proportion of (Agaricomycetes) Sebacinales was 83.4%, (Discomycete) Pezizales was 11.4%, (Leotiomycetes) Helotiales was 2.1%, unidentified was 2.3%. At Family level, Serendipitacea was 83.4% and Pezizaceae was 11.4%. At Genus level, Piriformospora was 83.4%. Analysis of Shannon index, Chao1, Observed species and Simpson showed that diversity of endophytic fungi in Bletilla striata were uniformly distributed.
自然条件下, 几乎所有的兰科植物都必须与菌根真菌形成共生关系才能完成正常的生长发育[1, 2]。1840年, Link首次证实了兰科植物根部有内生真菌, 然后不断有兰科植物与菌根真菌共生关系的报道。形成兰科菌根的真菌大部分属于担子菌(Basidiom-ycotina), 其无性态为丝核菌[3, 4]。近年来研究发现, 兰科菌根真菌还有少部分为子囊菌(Ascomycotina)和接合菌(Zygomycotina)[5]。Rasmussen[6]研究表明, 子囊菌也是兰科的主要菌根真菌, 而且越来越多的证据证实了这一观点。随着分子生物学在兰科菌根真菌领域的应用, 子囊菌开始受到广泛关注[3]。
白芨[Bletilla striata(Thunb.)Reichb.f. P.E.]为兰科(Orchidaceae)白芨属(Bletilla Reichb.f)植物, 是我国传统的中药材, 因其收敛止血、消肿生肌的功效而广泛应用于临床。内生真菌多样性的研究对深入了解白芨具有重要意义。目前, 我国主要以组织块培养法从兰科植物根部分离内生菌根真菌, 这种方法无法从中分离出所有的内生菌根真菌, 分离得到的真菌也无法确定是否为兰科的内生菌根真菌。分子生物技术的应用克服了传统方法的限制, 可以从基因水平上研究菌根真菌的多样性, 确定菌根真菌的群落特征。本试验以表面灭菌的白芨根为材料, 提取菌根总DNA, 采用高通量测序技术进行测序, 利用PEAR将成对的Reads进行合并过滤, 得到Clean Reads; 根据Clean Reads对样品进行序列可操作分类单元(Operational taxonomic units, OTU)、物种特异性和多样性分析, 以获得白芨根中内生真菌的多样性信息, 为进一步开展对白芨的研究提供科学依据和理论基础。
紫花三叉白芨新鲜根部, 2015年9月采自江苏句容。
1.2.1 白芨根组织处理及表面消毒
选择新鲜健康的白芨根, 用灭菌的去离子水冲洗干净, 然后分次浸入75%乙醇45 s, 2%次氯酸钠溶液浸泡消毒10 min。消毒完成后, 用灭菌的去离子水冲洗5~6次以彻底去除次氯酸钠; 最后超声波处理2次, 每次2 min, 以去除菌根表面残留的微生物DNA。用灭菌的滤纸吸干根表面的残留水滴, 然后用灭菌的解剖刀将根段切成0.5 mm左右的组织段。
1.2.2 菌根总DNA的提取
采用改良的CTAB法提取白芨菌根总DNA。具体步骤如下:
1)取0.5 g白芨根部组织, 液氮研磨, 磨碎物加入离心管; 2)向离心管中加入600 μ L预冷的CTAB-free, 冰浴10 min, 12 000 g离心5 min, 取上清液; 3)加入600 μ L预热的2% CTAB提取缓冲液, 65 ℃水浴45 min; 4)冷却至室温后加入等体积的饱和酚, 13 000 g离心15 min, 取上清液; 5)加入与上清液等体积的酚、氯仿和异戊醇的混和液(体积比为25:24:1), 13 000 g离心15 min, 取上清液; 6)加入与上清液等体积的氯仿、异戊醇的混和液(体积比为24:1), 13 000 g离心15 min, 取上清; 7)重复步骤6一次; 8)加入0.1倍体积的5 mol· L-1的醋酸钾和1倍体积的预冷异丙醇, 混匀后置-20 ℃ 30 min或过夜, 13 000 g离心10 min, 弃上清; 9)加入400 μ L预冷的70%乙醇使沉淀悬浮, 13 000 g离心5 min后弃上清液, 重复2~3次, 风干; 10)加入25 μ L的无菌高纯水, 4 ℃保存备用。
1.2.3 ITS rDNA文库构建
取10 ng DNA模板, 对目的区域真菌基因组转录间隔区1区(Internal transcribed spacer 1, ITS1)进行扩增。根据测序区域的不同, 选择对应区域引物。BITS (5'-ACCTGCGGARG GATCA-3')和B58S3 (5'- GAGATCCRTTGYTRAAAGTT-3')[7]。使用TaKaRa的EXtaq酶进行PCR扩增。
1.2.4 库检和高通量测序
使用Qubit 2.0稀释文库至1 ng· μ L-1, 对文库的insert size采用Agilent 2100进行检测, insert size符合预期后, 进行Q-PCR(Bio-RAD CFX 96荧光定量PCR仪, Bio-RAD KIT iQ SYBR GRN), 对文库的有效浓度进行准确定量。并对检测合格的文库进行高通量测序。
利用软件QIIME对Read 1和Read 2(分别从5’ 和3’ 端2个方向测序所得到的序列片段[8])拼接后的序列进行分析, 包括OTUs的提取、聚类分析、Alpha多样性分析等[9, 10, 11]。
OTU分析:采用Usearch61算法和UNITE真菌ITS数据库(http://unite.ut.ee/)对OTU进行划分, 并对OTU代表序列的分类信息进行确定, 在各个分类水平上统计各样本的群落组成, 并在真菌属的水平上计算真菌的相对丰度。
聚类分析:对样品的所有比对上参考数据库的序列总数(Total aligned reads)进行聚类, 默认以97%的一致性(identity)将序列聚类为OTUs。
Alpha多样性分析:用QIIME软件计算Shannon index、Chao1、Observed-species、Simpson指数。
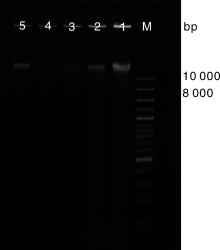
白芨菌根总DNA的琼脂糖凝胶电泳检测结果如图1所示。从图1可以看出, 1、2、5白芨菌根总DNA条带清晰完整, 符合后期进行菌根总DNA样品基因建库测序分析的要求。
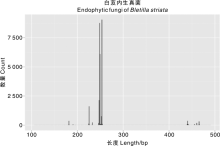
所测得的序列中能够成功合成的成对序列数(total assembled reads)为31 380个, 长度平均约256 bp, 拼接后序列长度分布如图2所示。
2.3.1 OTUs统计
本试验共获得33 255条序列(拼接成功的序列总数), 与数据库比对成功的序列总数为16 390, 可分为32个OTUs(97%的序列相似性)。
从表1可知, 序列数最多的OTU登录号是AF019636, 序列数达到13 667, 该菌株是Piriformospora indica。其次是登录号为FJ788739的Uncultured Pezizaceae, 序列数为1 858。序列数最少的有13个OTUs, 它们序列数量均为2个。
| 表1 OTUs统计分布表 Table 1 Statistical distribution table of OTUs |
2.3.2 OTUs丰度排秩分析
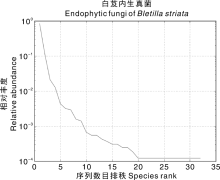
OTUs丰度排秩分析(Rank-Abundance)是依据每个OTU所含序列数的多少对OTUs进行排序, 白芨菌根样本里OTUs所包含的序列数分布情况可以通过Rank-Abundance直观地看出, OUTs按包含的序列数目排秩在20~32之间时, 曲线趋于平稳, 表明OTUs分布均匀(图3)。
2.3.3 白芨内生真菌饼图分析
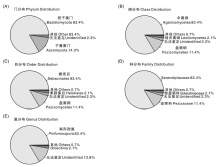
统计白芨菌根中的真菌, 根据百分比作饼图, 从门、纲、目、科、属不同分类水平进行统计(图4)。门分类水平上, 白芨内生真菌主要分布在担子菌门(83.4%), 其次是子囊菌门(14.3%), 还有2.3%是无法鉴定的。纲分类水平上, 内生真菌主要分布于伞菌纲(83.4%), 其次是盘菌纲(11.4%), 锤舌菌纲(Leotiomycetes)2.1%, 其他纲0.7%, 无法鉴定到纲的占2.3%。目分类水平上, 内生真菌分布于蜡壳菌目(83.4%), 盘菌目(11.4%), 柔膜菌目(2.1%), 其他目(0.7%), 无法鉴定到目的占2.3%。科分类水平上, 内生真菌主要分布于Serendipitaceae(83.4%)和盘菌科(11.4%)。从属的分类水平来看, 内生真菌主要为梨形孢属(Piriformospora)(83.4%)。
2.3.4 白芨内生真菌优势度指数分析
为更好地研究白芨菌根的优势内生真菌, 用公式di=Ni/Nt计算白芨菌根样品中每个真菌的优势度指数di; 其中, Ni表示物种i在样本中的个体数量, Nt则表示样品中所有物种的个体数量的总和。优势度指数可以体现某个物种在白芨菌根样品中所占的比例。经计算, 白芨菌根样品在门水平下的优势度指数为:担子菌门(0.834 1)、子囊菌门(0.142 6)、接合菌门(0.000 3)、无法鉴定(0.022 9)。
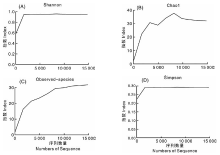
Alpha多样性可以反映样品内菌群的多样性, 包括菌种数目的均匀度以及菌种的类别丰富度。Alpha多样性越高即真菌种类越丰富, 真菌数目越均匀则表示此群落越稳定。本试验采用Shannon index、Chao1、Observed species和Simpson 4种指数对菌根样品中的内生真菌多样性进行分析(图5)。结果显示, 刚开始的测序量远不足以覆盖样品时, 4种指数曲线均出现直线上升, 随着测序数量的增加, 曲线斜率下降直至平稳, 说明白芨菌根样品测序合理, 可以反映白芨菌根中内生真菌的多样性。
自然条件下, 由于培养条件等方面的影响, 有85%~99.9%的微生物无法通过纯培养获得。因此, 常规方法不能分离和鉴定生活在组织内部的微生物, 而且分离获得的微生物也不一定是天然的优势菌群[12]。随着第二代测序技术的日趋成熟, 将Illumina高通量测序技术应用于兰科菌根中内生真菌研究, 可以克服组织块分离培养工作量大, 无法分离到菌根中不可人工培养的内生真菌, 以及传统分子生物学通量低等缺陷, 从基因组水平解析内生真菌的分布情况。
陈泽斌等[13]采用高通量测序技术研究了铁皮石斛叶片内生真菌的多样性, 获得的有效序列数为41 361, OTU数为33, 发现内生真菌裸胞壳属(Emericella sp.)的构巢裸壳孢属(Emericella nidulans)是铁皮石斛叶片的优势种群。陶刚[14]研究了贵州5个采样点的白芨根组织分离的内生真菌, 结果发现, 所分离到的内生真菌有26种属于子囊菌(占24.3%), 21种属于担子菌(占75.07%)。本试验采用Illumina高通量测序技术研究白芨根部的内生真菌, 证实了白芨根中确实存在内生真菌, 内生真菌分属于32个OTUs。从门的分类水平来看, 白芨内生真菌主要分布在担子菌门(83.4%)和子囊菌门(14.3%), 这和陶刚[14]的结果一致。从属的分类水平来看, 内生真菌主要分布于梨形孢属(Piriformospora sp.)(83.4%)。而袁宁[15]、陶刚[14]、陈晓芳等[16]、苏子敬等[17]虽然也分离到了担子菌门的内生真菌, 但未分离到梨形孢属真菌, 这可能与白芨的品种、采集地等因素有关[18], 生长在不同地方的同种植物, 可能有不同的内生真菌[18]。从种的分类水平来看, 白芨内生真菌主要属于印度梨形孢(Piriformospora indica), 因此, 印度梨形孢为白芨根部的优势真菌, 这也与席刚俊等[19]的鉴定结果一致。
(责任编辑 侯春晓)
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|