作者简介:邹琪琪(1989—),女,山东泰安人,硕士研究生,主要从事微生物遗传育种研究。E-mail:zouqiqisunny@163.com
为了构建甲基营养菌转录因子亚文库及筛选与甲醇脱氢酶启动子DNA相互作用的蛋白质,对甲基营养菌MP688的基因组和转录组数据进行分析,使用关键词对全基因注释信息和转录组结果进行搜索,找到相关基因的核苷酸序列,通过PCR获得目的片段并构建一系列转录因子亚文库,利用细菌单杂交系统筛选转录因子亚文库找到与甲醇脱氢酶启动子具有相互作用的调控因子。成功构建完成含有32个转录因子的亚文库,建立了甲基营养菌中细菌单杂交筛选方法,通过该方法筛选出2个与甲醇脱氢酶启动子具有强相互作用的转录因子基因。对于基因组已测序的细菌来说,构建转录因子亚文库筛选效率要优于构建基因组文库和cDNA文库,细菌单杂交系统能成功用于甲基营养菌,并能快速高效地筛选与启动子具有相互作用的转录因子。这一方法的建立,为阐明甲醇脱氢酶在甲基菌生长和代谢产物合成中的调控机制奠定了基础。
To construct the transcription factor sublibrary of methylotrophic bacteria and screen the proteins interacting with the methanol dehydrogenase promoter, we found many candidate genes by analyzing the genome and transcriptome databases of methylotrophic bacteria (MP688) in the use of some key words searching the genome annotation and transcriptome result, then amplified the target genes by PCR (polymerase chain reaction) and construct a series of transcription factor sublibrary. Finally B1H and EMSA (electrophoretic mobility shift assay) methods were employed to screen and verify the candidate factors. We had constructed a total of 32 transcriptional factor clones, and two transcriptional factors which have strong interaction with methanol dehydrogenase promoter were screened out. For bacteria having detailed genomic sequence information, the construction of the transcriptional factor sublibrary is better than the construction of the genomic library or cDNA library. B1H system can be successfully applied in screening the DNA binding proteins in methylotrophic bacteria. And the establishment of this method laid a solid foundation to clarify the regulatory mechanism of methanol dehydrogenase gene in the process of development and metabolic product in the methylotrophic bacteria.
甲基营养菌(Methylovorus)是一类能够利用甲醇等一碳碳源生存的微生物, 是研究一碳代谢的模式菌, 同时, 甲基营养型细菌代谢产物非常丰富, 是多种重要化合物工业生产的候选菌株[1]。其代谢产物吡咯喹啉醌(pyrroloquinoline quinone, 简称PQQ)是继烟酰胺核苷酸和黄素核苷酸之后发现的第三类氧化还原酶辅酶, 其具有多种生理功能, PQQ具有较强清除氧自由基的作用, 能够刺激动植物生长, 防治心脑疾病、肝损伤、促进神经保护等功能[2, 3, 4]。
甲醇脱氢酶(methanol dehydrogenase, MDH)MDH是甲基营养菌对甲醇利用和生物氧化的关键酶, 是一种以PQQ为辅基的醌蛋白, 其结构是由2个亚基组成, 为α 2β 2的四聚体。每分子酶含有2分子PQQ和2分子Ca2+, 且它们均位于酶活性中心处, 在酶的催化反应中起重要作用。在甲基营养菌中, 甲醇的氧化是通过甲醇脱氢酶进行的, 它催化甲醇氧化成甲醛。甲醇脱氢酶与甲醇及PQQ的结构和催化具有一定的联系, PQQ的构型变化是由甲醇脱氢酶酶促反应诱导的[5]。有文献报道, MDH对甲醇活力较低并不是酶本身的原因, 而是相应的底物和某些协助因子未能发挥最佳作用所造成的。李大攀等[6]敲除PQQ依赖的MDH基因后, MDH的活力及氧化甲醇的能力下降, 说明对甲醇的利用是多基因协同作用的结果。王冠芳等[7]提纯MDH进行活性检测及对底物催化专一性实验, 发现依赖PQQ甲醇脱氢酶对底物催化专一性差。李淼鑫等[8]从甲基营养菌中扩增甲醇脱氢酶(MDH)基因, 并在大肠埃希菌中表达, 检测其活性, 考察该基因对吡咯喹啉醌(PQQ)产生的影响, 该基因能够在大肠埃希菌中表达, 且表达产物具有生物活性; 提高MDH的表达水平对甲基营养菌PQQ的生物合成有一定影响。甲醇作为某些甲基营养菌株的惟一碳源, 对菌体生长和PQQ生物合成的影响较大, 甲醇脱氢酶是同化甲醇并为菌体生长提供能量的关键酶, 醌酶的表达水平对PQQ具有一定影响。本研究利用细菌单杂交技术, 筛选与甲基营养菌Methylovorus sp. MP688[9](中国普通微生物菌种保藏中心, 编号CGMCC4096)的甲醇脱氢酶启动子有相互作用的转录因子, 为阐明甲醇脱氢酶在甲基菌生长和代谢产物合成中的调控机制奠定基础。
1.1.1 菌株、质粒及引物
| 表1 菌株和质粒 Table 1 Strains and plasmids |
| 表2 本研究中使用的引物 Table 2 Primers used in this study |
1.1.2 培养基和培养条件
LB培养基:胰蛋白胨10 g· L-1, NaCl 5 g· L-1, 酵母提取物5 g· L-1, ddH2O 1 L调节pH至7.0, 用于大肠埃希菌的培养, 培养温度为37 ℃。
筛选培养基:琼脂粉1.5 g, ddH2O 76 mL, 调节pH至6.8~7.0, 121 ℃灭菌20 min, 琼脂冷却至70 ℃加入10 mL10× M9 salts, 混合均匀, 待混合物冷却至50 ℃时, 加入M9 Media Additive 13.5 mL, 氯霉素100 μ L, 链霉素100 μ L, 四环素50 μ L, 3-amino-1, 2, 4-triazole (3-AT) 1 mL。用于单杂交筛选, 培养温度为28 ℃。
1.1.3 主要试剂
质粒提取试剂盒、纯化PCR产物试剂盒均购自全式金生物技术有限公司, 限制性内切酶、T4连接酶购自Thermo公司。
大肠埃希菌质粒提取、PCR产物扩增、 DNA的限制性酶切与酶连反应、酶连产物和质粒的转化、DNA凝胶电泳等常规分子生物学实验操作参照《分子克隆实验指南》[17]。
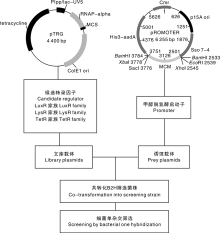
使用关键词“ regulator” “ sigma” “ sensor” “ two component” 对MP688全基因注释信息(GenBank编号:NC_014733.1)和转录组结果进行搜索, 找到相关基因的核苷酸序列即为候选转录因子序列。我们分类查找到LuxR、LysR、TetR家族特异性转录因子, 以MP688菌株的基因组为模板PCR扩增得到转录因子共计32个。将各转录因子与文库载体pTRG进行酶切位点分析, 查找合适的酶切位点BamHⅠ 、EcoRⅠ 或XhoⅠ , 进行重组质粒的构建, 得到的一系列质粒即构成了甲基营养菌Methylovorus sp. MP688的转录因子亚文库。
1.4.1 共转化菌株的构建
由于B2H validation strain感受态细胞的质粒中存在卡那霉素抗性基因, 文库载体含有四环素抗性基因, 而报告质粒中存在氯霉素抗性基因, 因此在含3种抗生素的培养基上生长的菌落可以视为已经转入了重组质粒与报告质粒, 即共转化成功。将文库载体和重组报告质粒各8 μ L共转化到50 μ L B2H validation strain感受态细胞中, 涂布到含卡那霉素、四环素、氯霉素的LB固体培养基上, 37 ℃倒置培养12~14 h, 长出的菌落即为共转化菌株。
1.4.2 点种及培养
接种所构建的共转化菌株单菌落至LB液体培养基(卡纳霉素、四环素、氯霉素), 37 ℃过夜培养。将筛选平板与对照保存平板(添加有3种抗性的LB平板)置于超净工作台中, 适当打开平皿盖以吹干培养基表面的多余水分, 并且保证培养基表面不会过于干燥, 取所接种的共转化子菌液2 μ L分别点种于筛选平板及对照保存平板, 点种后将平板正向置于30 ℃培养箱中约30 min, 待所点菌液完全被培养基吸收后, 将平板倒置避光培养。
1.4.3 筛选平板上菌落生长情况的观察
将筛选平板与对照平板倒置于30 ℃培养箱中培养约12 h, 取出观察并记录菌落生长情况, 主要包括菌落是否生长、菌落形态、相对生长趋势等。平板继续倒置于30 ℃培养箱培养, 每隔12 h左右按照相同标准观察并记录筛选平板, 培养至第3天为止。启动子与转录因子相互作用的强弱主要表现在菌落的生长情况, 因此可根据菌株生长情况判断各转录因子是否与甲醇脱氢酶启动子发生相互作用及相互作用的强弱。
1.4.4 阴性对照自激活验证及3-AT浓度确定
将pTRG空质粒和克隆有甲醇脱氢酶启动子序列的pROMOTER质粒共转化到报告菌株B2H validation strain中, 在筛选培养基中观察报告基因的表达状况。若有菌落生长则在培养基中添加不同浓度的3-AT, 直至培养基上无菌落生长。
1.4.5 甲基营养菌中DNA-蛋白质相互作用的阳性对照的筛选
以模式菌Methylobacterium extorquens AM1的AM1_glyA、AM1_qsr基因为参考, 与Methylovorus sp. MP688全基因组进行同源分析, 得到同源性较高的基因mpq_0872、mpq_1945, 并查找RNA聚合酶的不同亚基得到MPQ_0345、MPQ_2317、MPQ_0345CTD的相关基因序列, 以此作为阳性对照的候选因子。
1.4.6 MPQ_1945阳性对照蛋白的构建与表达
将PCR扩增的mpq_1945基因经NdeⅠ 和BamHⅠ 双酶切, 选择表达载体pET-15b, 用NdeⅠ 和BamHⅠ 线性化此载体, 然后用T4连接酶连接双酶切的mpq_1945基因片段及线性化pET-15b载体, 构建mpq_1945的表达质粒。再转化至E. coli BL21感受态细胞中。挑选正确的转化子菌株接种至含1 μ g· mL-1氨苄抗生素培养基中, 37 ℃过夜培养后以1%接种量转接该菌液到100 mL培养基中, 培养至对数生长期(D600=0.4)后加入IPTG至终浓度依次为0.10、0.25、0.50 mmol· L-1, 20 ℃诱导过夜培养, 离心菌液收集表达菌体, 所得菌体经超声波破碎(Pulse on 3 s, off 6 s)后, 12 000 r· min-1离心30 min, 取上清液用镍柱亲和层析法纯化蛋白, 10% SDS-PAGE检测目的蛋白的表达和纯化情况。
1.4.7 凝胶阻滞实验(EMSA)
将mpq_0872基因启动子的DNA片段和MPQ_1945蛋白以一定比例添加至1.5 mL离心管, 加入10× 反应缓冲液(20 mmol· L-1 Tris-HCl pH 7.5, 100 mmol· L-1 NaCl, 2 mmol· L-1 EDTA, 0.5 mmol· L-1 MgCl2)混匀, 4 ℃静置孵育2 h, 体系见表3。制备6.5%非变性聚丙烯酰胺凝胶, 体系总体积10 mL, 含5× TBE 1mL, 30%聚丙烯酰胺2.2 mL, 80%甘油80 μ L, 10%过硫酸铵90 μ L, 四甲基乙二胺10 μ L, 去离子水6.62 mL。加样前先在预冷的0.5× TBE 缓冲液中120 V预电泳10 min, 电泳完毕后冲洗加样孔。将混合样品进行点样电泳, 将电泳槽置于冰上或4 ℃环境中, 恒压100 V进行电泳。电泳结束后用溴化乙锭染色。
| 表3 EMSA反应体系 Table 3 EMSA reaction system |
LuxR、LysR、TetR家族转录因子在细菌中普遍存在, 并与多种次级代谢产物合成密切相关, 通过对甲基营养菌Methylovorus sp. MP688全基因组及转录组测序结果的分析, 并对LuxR、LysR、TetR家族的32个转录因子基因分类整理, 结果表明, 有11个转录因子基因属于LuxR家族, 有11个转录因子基因属于LysR家族, 有9个转录因子基因属于TetR家族。针对LuxR家族、LysR家族转录因子基因设计特异性引物, 扩增目的因子并与pTRG质粒克隆, 构建重组质粒, 经酶切验证, 构建成功的LuxR转录因子亚文库有6个, 分别为LuxR1、LuxR2、LuxR5、LuxR6、LuxR7、LuxR8; LysR转录因子亚文库有5个, 分别为LysR1、LysR2、LysR4、LysR5、LysR6。
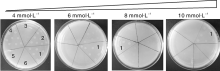
由于转录因子能够与RNA聚合酶的α 亚基融合表达, 将文库载体及报告载体共转化到宿主菌株中后, 若调控蛋白和启动子存在相互作用, α 亚基便会牵引RNA聚合酶使其牢固地结合在目的启动子区, 从而激活之后的报告基因his3和aadA的转录表达。为了验证在没有启动子时该系统是否具有自激活现象, 设计阴性对照共转化菌株的单杂交筛选。结果表明, 阴性对照菌株在筛选平板上生长, 证明有自激活现象, 在培养基中添加2~4 mmol· L-1的3-AT能够抑制其自激活。阴性对照在浓度为2、3 mmol· L-1的3-AT下可以生长, 当浓度增至4 mmol· L-1时对照菌的生长被抑制, 因而确定3-AT的最适筛选浓度为4 mmol· L-1, 在此浓度下, 能够确保非特异性自激活被充分抑制, 且抑制剂浓度最低。
由于Methylobacterium extorquens AM1和Methylovorus sp. MP688均为甲基营养型菌株, 具有同源性。为了寻找甲基营养菌中具有相互作用的阳性对照, 我们以与AM1具有相互作用的转录因子QSR为参考, 对Methylovorus sp. MP688基因组进行基因分析比对, 将与AM1具有同源性的基因作为阳性候选基因并构建共转化菌株。细菌单杂交筛选中, 以4 mmol· L-1 3-AT浓度为基准, 设定3-AT的浓度梯度为4、6、8、10 mmol· L-1。对其进行细菌单杂交筛选, 结果表明, 随着3-AT浓度的增加, 含不同转录因子的菌株长势发生变化, 如图2所示, 在4 mmol· L-1 3-AT浓度下, AM1_QSR、MPQ_0345、MPQ_0345CTD、MPQ_1945正常生长; 浓度为6 mmol· L-1时, AM1_QSR、MPQ_0345、MPQ_1945能够生长; 浓度为8 mmol· L-1时, AM1_QSR、MPQ_1945继续生长; 浓度为10 mmol· L-1时, AM1_QSR、MPQ_1945仍能正常生长, 最终确定MPQ_1945作为甲基营养菌细菌单杂交筛选模型的阳性对照。在此浓度下, 能确保非特异性自激活被充分抑制, 而含有MPQ_0872和MPQ_1945相互作用的菌株生长良好。
阳性对照蛋白MPQ_1945进行IPTG浓度梯度优化, 最终在IPTG浓度为0.25 mmol· L-1时得到可溶性蛋白, 用镍柱亲和层析进行纯化得到目的蛋白, 如图3所示。
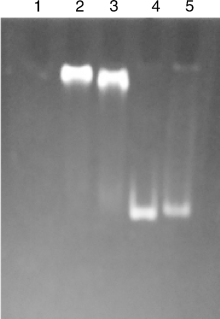
细菌单杂交系统筛选出的DNA-蛋白质间相互作用的蛋白是否可靠, 我们结合凝胶阻滞实验来证实。将筛选的阳性MPQ_1945蛋白和mpq_0872基因的启动子进行EMSA, 反应体系见表3, 经EB染色后, 由于体系1没有DNA, 所以EB染色没有条带, 体系2、3同时具有DNA和蛋白质, 两者结合后电泳时比无蛋白质的DNA游离的慢, 因此EB染色滞后。体系4只有DNA, 作为阳性对照DNA经EB染色后呈游离带, 体系5同时含有DNA和蛋白质, 然而该蛋白质是甲基菌的无关蛋白, 以体系5作为阴性对照, 经EB染色后呈游离带。EB染色结果如图4所示, 最终确定细菌单杂交系统能在甲基营养菌中应用并具有可靠性。
2.6.1 甲醇脱氢酶启动子序列分析
通过生物信息学预测(http://linux1.softberry.com/berry.phtml)mpq_0771基因前有一个得分高的强启动子, 如图5所示。该启动子的-10区和-35区以及可能的转录因子结合位点也标识出。因此我们设计PCR引物扩增了540 bp包含这部分功能区的片段, 将构建到pROMOTER的报告基因前, 通过细菌单杂交筛选这些DNA位点的结合蛋白。
2.6.2 甲醇脱氢酶启动子细菌单杂交筛选
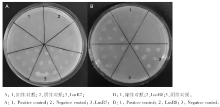
通过观察和记录含有不同浓度抑制剂3-AT的筛选培养基中共转化子的生长状况, 可知随着3-AT浓度的增加各共转化菌株长势不同, 部分长势良好, 部分长势较弱, 说明与甲醇启动子有不同程度作用结果。筛选结果中无相互作用的有1个, 弱相互作用的有2个, 中等作用的有6个, 结果见表5, 当3-AT浓度增加至15 mmol· L-1时阳性对照菌生长状态趋于下降, 而LuxR7、LuxR8的共转化菌株长势依旧良好, 结果见图6, 表明LuxR7、LuxR8转录因子与报告载体上的甲醇脱氢酶启动子具有很强的相互作用, 相关结合蛋白的信息见表6。
| 表5 细菌单杂交筛选 Table 5 The bacterial-one-hybrid screening |
| 表6 结合蛋白信息 Table 6 The information of binding proteins |
本实验室构建的细菌单杂交系统从自身系统的验证及将已知的转录因子和启动子的相互作用作为对照来寻找最佳筛选条件, 最终确定3-AT最适筛选浓度为4 mmol· L-1。由于细菌单杂交存在假阳性和假阴性的缺陷, 我们用EMSA来进一步验证所筛选的结果, EMSA实验结果与单杂交结果一致, 证明了细菌单杂交系统在甲基营养菌高通量筛选中的可行性[10, 11, 12, 13]。
转录因子也称为反式作用因子, 是一类细胞核内的蛋白质因子, 通过与顺式作用元件和RNA聚合酶的相互作用来调节转录的活性。由于细菌mRNA分离困难, 构建细菌cDNA文库一直是学术界难点, 而通常细菌基因组文库的建立又由于采用基因组随机断裂的方法, 存在着一些缺点, 如:插入片段的随机性和定向性差等缺点, 因此采用这种方式构建细菌cDNA文库对其文库的覆盖度和效率要求较高。本实验基于甲基营养菌的全基因组序列, 对其进行分析找到和转录调控有关的基因序列, 包括LuxR、LysR、TetR家族转录因子总计32个, 最后通过逐一扩增转录因子基因, 来构建转录因子亚文库。从而使文库的库容量和效率大大增加, 同时由于筛选范围的缩小, 使筛选目标更具定向性。比如, 甲基营养菌是合成PQQ的主要菌株, pqqABCDE等PQQ合成模块基因虽然在不同菌株中存在差异, 但这些基因的转录受到严格的调控[14, 15], 目前关于PQQ合成调控的研究较少。本文建立的方法可用于高通量筛选PQQ合成模块基因的启动子结合蛋白, 最终阐述甲基营养菌PQQ合成调控机制。
在基因的表达调控中, 参与调控的转录因子是各种蛋白质, 而核酸和蛋白质又是构成生命体最为重要的两类生物大分子, 因此研究蛋白质-蛋白质、DNA-蛋白质等生物大分子之间的相互作用对于人们在分子水平上深入理解各种生物学过程至关重要[16]。本实验对细菌单杂交系统进行优化使其成功应用于甲基营养菌, 并利用细菌单杂交技术的独有的优点, 筛选转录因子亚文库, 成功从32个转录因子中筛选出与甲基营养菌甲醇脱氢酶启动子相互作用的蛋白, 共计11个。这些与甲醇脱氢酶启动子有相互作用的蛋白又表现出强弱程度的不同。为进一步阐明甲醇脱氢酶在甲基菌生长和代谢产物合成中的调控机制奠定了基础。同时构建成功的文库也能够在其他基因转录因子筛选中得到应用, 为构建PQQ高产菌株及诠释PQQ合成基因的转录调控机制提供了理论依据。
(责任编辑 张 韵)
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|