作者简介:汤婷(1993—),女,安徽铜陵人,硕士研究生,研究方向为微生物。E-mail:yyqxtt1993@163.com
花椰菜花叶病毒35S启动子(CaMV35S)是植物基因工程中应用最广泛的启动子之一,它在转基因植物的安全评价及检测研究中具有重要意义。了解CaMV35S启动子的起源及其发展情况,是开展转基因安全评价和检测的前提。文章对CaMV35S启动子的发展历史及其结构、功能作了简要描述,分析总结了CaMV35S启动子在转基因作物中的应用情况和目前对其相应的检测方法。针对目前在转基因作物中检测CaMV35S启动子存在的问题,认为今后转基因相关作物的序列信息及检测数据需要交流和共享,同时各个检测机构或实验室之间需要展开数据共享与共同验证,从而建立针对CaMV35S启动子的相对统一的标准检测方法。
CaMV35S promoter is one of the most important promoter that widely used in plant genetic engineering. It plays a key role in the safety evaluation and detection of transgenic plants. Understanding the origin and development of CaMV35S promoter is the prerequisite for carrying out safety assessment and detection of transgenic plants. In this review, history of CaMV35S promoter and its structure and function were briefly described. Then, application of CaMV35S promoter in transgenic crops and the corresponding detection methods were analyzed and summarized. Finally, aiming at the problems existing in detection of CaMV35S promoter in transgenic crops, it was proposed that in future studies, sequence information and detection data of related transgenic crops should be communicated and shared, meanwhile, data sharing and joint verification should be carried out among various testing institutions or laboratories, so as to establish a relatively uniform standard detection method for CaMV35S promoter.
从1996年美国最早开始商业化生产和销售转基因作物开始, 转基因作物的商业化在争论中得到迅速发展。目前, 共有40个国家开展了转基因作物的商业化种植。而在转基因作物中, 外源目的基因被整合进宿主细胞时, 1个能够高效启动目的基因表达的启动子是必不可少的。启动子是RNA聚合酶能够识别并与之结合, 从而促使基因转录的一段DNA序列, 是决定基因表达时间、部位、强度等的主要调控元件, 是完成基因表达的表达元件中至关重要的一部分, 通常位于基因上游。一个典型的启动子包括CAAT-box和TATA-box, 它们分别是依赖DNA的RNA聚合酶的识别和结合位点, 一般位于转录起始位点上游几十个碱基处。在核心启动子上游通常会有一些特殊的DNA序列, 即顺式作用元件, 转录因子与之结合从而激活或抑制基因的转录。一旦RNA聚合酶定位并结合在启动子上即可启动基因转录, 因此, 启动子是基因表达调控的重要元件, 它与RNA聚合酶及其他蛋白辅助因子等反式作用因子的相互作用是启动子调控基因转录的实质。根据启动子的转录模式可将其分为3类:组成型启动子、组织或器官特异性启动子和诱导型启动子[1, 2]。其中, 组成型启动子能在所有组织中都启动基因表达, 具有持续性, 不表现时空特异性, RNA和蛋白质表达量也是相对恒定的。从结构上看, 大多数组成型启动子转录起始位点上游几百个核苷酸处, 都存在核苷酸序列TGACTG[3]。基因工程中, 花椰菜花叶病毒(CaMV35S)启动子被广泛地使用, 其能够在许多植物物种的几乎所有发育阶段及所有组织中高效表达, 被广泛应用于转基因植株, 如玉米、棉花、大豆、番茄等的构建[4]。
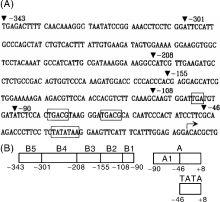
花椰菜花叶病毒(Cauliflower mosaic virus, CaMV)是一种感染十字花科成员的DNA病毒。该病毒长度大约8 kb, 它的完整核苷酸序列已经被确定[5]。在病毒的生命周期中, 35S启动子开启病毒DNA的转录, 产生一个8 kb的转录本, 称为35S RNA[6]。35S启动子起始于CaMV 35S RNA转录起始点-941至+208的Bg l Ⅱ 片段, 在位于CaMV 35S DNA中的-46至-105区段存在增强子序列, 该启动子包括TATA盒、倒转重复序列和CAAT盒、核心序列4个部分(图1-A)为CaMV35S序列[7]。1985年, Odell等[8]首次展示了CaMV35S的结构和功能, Jefferson等[9]和Sanders等[10]利用CaMV35S启动子测量了其在转基因植物叶茎根花中对报告基因的转录促进能力。Odell等[8]认为, CaMV35S启动子启动基因转录要依赖其周围临近序列, 包括一个TATA框, 然而, 转录的速度却由其上游大约300 bp的分散DNA 序列决定。Simpson等[11]认为, 这个区域是一个增强子(能够提高转录速率的DNA序列)。Odell等[8]的研究表明, -343至-46的上游片段是35S启动子的主要部分。在之后的研究中, Fang等[12]发现35S启动子可以划分为2个区域:从-90 bp至+8 bp为A区域, 主要负责基因在胚根、胚乳及根组织内表达; 区域B(-343至-90 bp)主要控制基因在胚的子叶、成熟植株的叶组织及维管组织内表达。图1-B展示了CaMV35S序列划分的不同区域。在B区域内的增强子序列可以提高表达水平, 若是删除-343至-208区域, 基因的转录强度减少50%。如果35S启动子中存在2个B区将使35S启动子活性提高10倍, 对异源启动子也有作用, 但B区对某些启动子起表达阻遏因子作用。
20世纪80年代初, Chua和洛克菲勒大学的合作者在感染了花椰菜花叶病毒(CaMV)的植物中分离出了一种启动子, 该启动子负责花椰菜花叶病毒(CaMV)的整个基因组的转录表达, 它的沉降系数是35S, 因此, 这个启动子被命名为CaMV35S启动子, 它是目前广泛使用的组成型启动子之一。已被实验证实的组成型病毒启动子有2种, 分别为19S和35S。其中, 19S启动子负责转录CaMV病毒部分基因, 但是没有35S应用广泛。35S RNA, 除了作为翻译的模板, 它的直接末端重复序列, 通过逆转录酶也可以作为病毒DNA合成的中间产物[13]。另外, Guilley等[6]研究表明, 在受感染的细胞中, 病毒快速复制需要大量的35S RNA, 35S RNA的高水平生产证明了这个启动子的强度。Kuhlemeier等[14]根据35S转录起始位点的信息及克隆CaMV序列的可用性, 鼓励使用35S启动子作为植物基因表达研究的模型系统。Morelli等[15]和Nagy等[16]对转基因烟草细胞或转基因矮牵牛花[17]相对转录水平的定量测定显示, 35S启动子比NOS启动子强至少30倍。同时, CaMV35S启动子具适度的天然启动能力。因此, 在转基因植物中, CaMV35S启动子被大量地用于外源基因调控。实验表明, 将包含35S上游序列400~1 000 bp的CaMV片段插入烟草[18, 19]或是矮牵牛花[17]的基因, 可以显示出活性。此外, 启动子还可以在几个双子叶植物和单子叶植物的原生质体中表达[20, 21, 22, 23]。
在植物转基因研究中, 使用天然启动子往往不能得到满意的结果, 尤其是在特异表达或诱导表达时, 表达水平不够理想。因此, 选择合适的启动子或对启动子进行改造, 是增强外源基因特异表达的重要手段。Lv等[24]将35S最小启动子结合在特异表达启动子GRPp的5'端形成双向启动子, 这些双向启动子能够指导GUS和GFP基因在所有独立的转基因烟草的维管特异表达。转基因植物的维管特异性双元双向启动子可用于同时调控某些维管特异性功能基因的表达。这一方法还可用于其他植物中极性启动子的生物技术研究。张秀春等[25]利用植物防御基因中的病原诱导响应元件和最小35S启动子(-62至+1), 人工合成了启动子SAR, 并以GUS基因为报告基因, 在转基因拟南芥中研究人工启动子的表达特性。结果表明, SAR启动子在子叶、毛刺、根茎交接处和根系中优势表达, 在老叶的表达量高于幼叶, 说明SAR启动子具有组织和发育时期表达特异性。彭舒[26]将筛选得到的F-box、GST1-box、S-box、W-box作用元件进行随机串联后与35S最小启动子进行组合, 成功构建了人工病原物诱导型启动子和植物表达载体。Xie等[27]将CaMV35S启动子上游反向连接了51 bp的Pmini启动子, 改造后的这个启动子具有双向表达功能, 并且2个方向表达强度相同, 同时改造后的双向启动子在拟南芥中的遗传性和稳定性表现良好。
自1986年世界上第1例转基因作物问世以来, 转基因作物迅猛发展[28]。转基因作物具有抗虫、抗病、抗逆、高产、优质等优点, 使其得到世界范围的广泛关注。根据全球农业生物技术应用咨询署(ISAAA)提供的统计资料, 2016年, 美国、阿根廷、加拿大等28个以上国家种植了大豆、玉米、棉花、油菜、马铃薯等转基因作物。虽然, 转基因技术给人们带来了巨大的经济效益, 但转基因作物及其食品的环境释放安全性及食用安全性也存在巨大争议。为保护贸易、明确消费, 各国政府均对安全监管提出了更高的要求[29]。作为转基因作物安全性监管的支撑, 转基因产品检测技术也成为研究的热点。常用的转基因检测方法主要有基于蛋白质的检测技术和基于DNA的检测技术。如酶联免疫吸附检测(ELISA)、聚合酶链式反应(PCR)、Southern杂交、基因芯片和DNA电化学传感器技术等。其中, 以PCR为主的基于DNA序列的转基因检测方法是普遍接受的转基因检测技术, 其有能力从高度加工的材料中扩增特定的DNA片段, 其主要依据是转基因作物中特异性外源DNA片段。用于植物转化的外源DNA片段可以分为通用元件、目的基因、外源载体序列3大类, 根据不同的外源DNA片段, 检测策略可以分为4个层次:筛选检测、基因特异性检测、载体特异性检测和转化事件特异性检测[30]。其中, 最常用的一个元件就是CaMV35S启动子。数据表明, 2013年, 有来自27种不同物种的336种转基因作物被商业化[31], 随着越来越多的转基因作物进入田间试验, 这一数字还在不断增加, 在此过程中, 仅开发了部分品种的转化事件特性检测方法。一般来说, 在转基因检测过程中对所有可能发生的转化事件进行PCR检测是不可行的。一种常见的做法是对许多转化事件常见的目标进行筛选, 而CaMV35S启动子就是这样的一个常见目标。运用转基因作物数据库(http://ceragmc.org/index.php?action5gm_crop_database)中可用的转基因信息, 对转基因作物中CaMV35S和TNOS进行了初步的统计分析, 结果见表1。
| 表1 转基因作物中含有的CaMV35S和TNOS数据统计 Table 1 Data statistics of CaMV35S and TNOS included in transgenic crops |
由表1可知, 经过批准的商业转基因转化事件的65.7%(67/102)包含了CaMV35S启动子, 53.49%(55/102)包含NOS, 以及81.4%(83/102)包含任一个或者两者。在重要的商业转基因作物中, CaMV35S和TNOS存在比例更高。在转基因玉米(Zea mays)的28个商业转化事件中, 只有1个没有这2个元件, 而在16个商业的油菜籽(Brassica napus)中, 只有2个例外。一些转基因的转化事件, 如转基因玉米BT11、T25和转基因棉花MON531, 它们的改造结构中包含了2个CaMV35S序列。
由于CaMV35S启动子对转基因作物的检测具有重要意义, 因此, 研究者们建立了大量的转基因筛选方法, 其中一些方法已被国际标准化组织(ISO)、欧洲联盟(欧盟)、中国及其他国家和地区采用, 并作为转基因检测的标准方法[32, 33, 34, 35, 36]。表2列举了来自不同文献和检测标准的25种检测CaMV35S启动子方法[37], 分别命名为M1~M25。其中, M1被ISO 2156915通过, M13被ISO 2157032通过, M1、M14、M11通过了中国的国家标准, M1、M2、M15、M16通过了中国的工业标准, 而M1、M4、M12、M14被欧盟数据库收集, 用于转基因成分分析。这25种方法包含了10种传统PCR方法和15种实时定量PCR方法。用于实时定量PCR方法的部分引物对, 如M1、M3、M4、M5、M12和M14, 也可以用于常规的定性PCR检测。另外, 表2中也列出了每种方法的引物/探针相对于CaMV序列的位置和扩增子的序列大小。CaMV35S启动子是GMO中最常修改的元件之一, 在不同的基因和载体上, 由于来源不同的菌株或在载体构建过程中的修饰或突变, CaMV35S启动子序列可能存在差异[34]。由寡核苷酸序列的比对结果可知, 转化事件176、BT11、T25、MON810和DLL2517中的CaMV35S启动子序列来源于CaMV基因组[38](图2)。本课题组收集了玉米、大豆等一些常见转基因作物的不同转化事件中的CaMV35S序列, 利用Vector NIT软件将这些序列进行比对, 结果显示, 这些转化事件中CaMV35S序列存在一定差异, 所有比对的CaMV35S序列都不完全一致(图2)。因此, 许多基于CaMV35S启动子的检测方法可用于转基因检测, 但是只有部分方法通过了必要的验证过程和实验室内对少量的转基因转化事件方法验证。Morisset等[39]在TC1507玉米的CaMV35S序列中发现了一个单核苷酸多态性(SNP), 检测引物/探针的结合位点出现SNP导致CaMV35S扩增效率不高。国际生命科学研究所(ILSI)请求100多个实验室使用CaMV35S和TNOS来检测转基因作物, 一些实验室在CaMV35S测试中遇到了方法学缺陷, 如灵敏度低、重现性低、误报或否定。Holden等[40]用8种玉米参考材料测试了5种基于CaMV35S的方法的适用性, 结果表明, 2种方法在一些测试材料中存在线性回归参数的缺陷和多重PCR扩增的缺陷。如果在转换载体或育种过程中, 测试样品携带的CaMV35S序列被改变, 那么该方法的差异性可能会导致不同的测试结果。ILSI的调查显示, 大部分参与实验的实验室都希望有一种标准化的方法, 这种方法可以产生一致的检测结果, 而且实验的重现性更好。综合比较现有方法, 还没有最优的方法可用。
| 表2 用于检测CaMV35S启动子的定性和实时定量PCR系统中使用的引物和荧光探针 Table 2 Primers and fluorescent probes used in detection of CaMV35S promoter in qualitative and real-time quantitative PCR systems |
目前, 针对CaMV35S的转基因成分快速筛查检测, 迫切需要找到不同CaMV35S序列中的保守序列, 根据这这段保守序列, 设计相应的引物进行检测, 能够同时满足不同转基因作物中CaMV35S的检测需要, 避免由于CaMV35S序列差异而造成的不同检测结果, 从而建立检测CaMV35S的统一方法。
转基因食品及其安全问题近年来已经成为全球关注的热点[41, 42, 43], 世界各国纷纷出台政策, 要求对转基因产品进行限量标识。我国于2001年和2002年先后发布了《农业转基因生物安全管理条例》《农业转基因生物标识管理办法》等配套管理办法, 明确规定对进入我国流通市场的转基因生物及产品实施标识制度。目前, 基于DNA的PCR技术是转基因成分检测的最主要手段。根据检测目标及特异性的差异可将PCR技术分为:筛查检测、基因检测、构建检测及转化体特异性检测。其中, 筛查检测的目标主要是转基因作物中常用的一些共用元件, 如启动子、终止子及标记基因, 虽然其特异性不强, 但因为针对的是共用元件的检测, 其检测的覆盖率最高。在日常检测中, 面对大量的检测样品, 筛查检测是最主要的前期检测方法, CaMV35S启动子是转基因成分筛查检测的主要元件之一。
目前, 检测CaMV35S启动子的PCR方法中存在以下问题:首先, 针对CaMV35S启动子的检测引物繁多, 只有一部分是来自ISO标准, 或相应国家或地区的标准, 经过了实验室研究和实验之间的验证过程, 多数引物是各个实验室内部开发的; 这些引物针对的CaMV35S启动子序列不同, 所以会产生不一致的检测结果, 从而给转基因成分检测的结果及后续的监测和监管造成巨大影响; 尤其是处在全球贸易的今天, 针对进出口产品的检测结果差异会造成一定的贸易争端。其次, 面对这些多而杂的检测方法, 给检测者造成了选择困难; 针对不同的检测样品, 在众多方法中选择一种简便准确的检测方法具有一定的难度。所以, 建立标准化的CaMV35S启动子检测方法很重要。但是, 由于目前检测机构或研究室之间的实验数据还没有做到全面共享和互用。目前, 还没有一个数据库包含了目前所有转基因作物的全部序列信息, 包括CaMV35S启动子序列信息, 因此很难建立一种标准化的检测方法。在今后的研究中, 针对CaMV35S启动子检测方法的研究, 相关转基因作物的序列信息及检测数据需要进行交流和共享, 从而使检测人员了解相应转化体中CaMV35S启动子的应用情况, 以及设计的检测引物的覆盖率。同时各个检测机构或实验室之间需要展开数据共享与共同验证, 建立针对CaMV35S启动子的相对统一的标准检测方法。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|