作者简介:张旭(1994—),男,四川乐山人,硕士研究生,主要从事果树栽培理论与技术研究。E-mail: 806174404@qq.com
柑橘果实粒化是成熟期以及贮藏期容易发生的一种生理性病害。木质素的积累与柑橘粒化的形成密切相关,汁胞木质化作为柑橘果实粒化过程中最明显的现象之一,备受果树研究者们的关注,并取得了一定的研究进展。本文以木质素的生物合成及其调控方式作为研究柑橘果实汁胞粒化的切入点,对木质素生物合成途径中的关键酶基因以及相关转录因子的研究进展进行综述,并对木质素合成途径调控柑橘果实粒化形成机制中存在的问题进行总结以及今后的研究方向进行展望,以期为柑橘果实粒化的后续研究提供借鉴。
The granulation of citrus fruits is a physiological disease that is easy to occur in fruit ripening and postharvest storage. The accumulation of lignin is closely related to the formation of granulation of citrus fruits. The lignification of juice sacs is one of the most obvious phenomena in the process of granulation of citrus fruits. It has attracted the attention of fruit tree research scholars and achieved certain research progress. Therefore, this paper will use lignin biosynthesis and its regulation as the entry point to study the granulation of citrus fruits, review the research progress of key enzyme genes and related transcription factors in lignin biosynthesis pathway, and the problems in the mechanism of lignin synthesis in the regulation of citrus fruit granulation were summarized and the future research directions were prospected in order to provide reference for the follow-up research on citrus fruit granulation.
柑橘果实易在成熟期和采后贮藏的过程中发生汁胞粒化现象, 表现为汁胞变硬, 颜色变白, 风味变淡, 导致果实品质严重下降, 甚至丧失商品价值。相关研究表明, 在沙田柚、红肉蜜柚和伦晚脐橙的粒化汁胞中观察到明显的木质化现象, 粒化汁胞中存在显著的木质素积累现象[1, 2]。利用低场核磁成像对伏令夏橙粒化汁胞进行研究发现, 随着汁胞粒化程度的加剧, 结合水数量增加, 木质素含量与粒化程度呈显著正相关[3]。木质素作为植物细胞壁的主要组成部分, 与纤维素、半纤维素、果胶等物质交叉联接。木质素的合成能够对植物细胞起重要的保护作用, 增强植物细胞抵抗病害和逆境的能力。但就园艺商品而言, 木质素的积累会造成果实品质的下降, 对园艺产业的发展是不利的。木质素生物合成的研究已经持续了很长时间, 由于其合成途径是一个非常复杂的过程, 目前关于木质素生物合成的机理仍在不断探索中。
木质素是由苯丙烷单体及其衍生物聚合形成的复杂酚类聚合物, 依据不同的苯丙烷单体可将木质素分为由香豆醇合成的对-羟基苯基木质素(H)、由松伯醇合成的愈创木基木质素(G)、由芥子醇合成的紫丁香基木质素(S)三种类型[4]。不同的植物含有的木质素类型和组成比例不同, 果树上的研究发现, 砀山酥梨果实中主要以G、S型木质素为主, 且G/S的比值会影响木质素的降解, 从而决定果实中石细胞含量[5]。琯溪蜜柚粒化汁胞中发现木质素主要为G型木质素[6]。
近年来, 随着木质素生物合成途径的不断深入研究, 对其合成途径的调控网络也在不断地更新。如图1所示, 植物经光合作用生成的同化物通过莽草酸途径生成苯丙氨酸等芳香族氨基酸; 苯丙氨酸通过苯丙烷途径生成羟基肉桂酸及其辅酶A酯类化合物; 羟基肉桂酸及其辅酶A酯类化合物通过木质素合成特异途径生成木质素单体, 木质素单体最终聚合形成木质素[7]。至今在莽草酸途径中还未能准确找到木质素生物合成的调控靶点, 但在苯丙烷代谢途径和木质素合成特异途径中发现了调控木质素合成的关键酶和相关转录因子[8]。因此, 这两个阶段的研究成为了学者们的研究重点。近年来, 利用生物技术手段, 对参与这两个阶段的关键酶进行了克隆并通过转基因试验证实了这些关键酶在木质素生物合成过程中的作用。但这些酶是单一地参与木质素的生物合成, 还是与上下游的关键酶形成调控网络参与到木质素生物合成, 目前还没有形成明确的定论[9]。
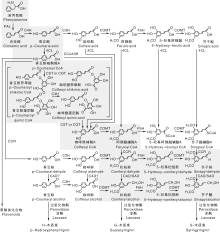 | 图1 木质素合成途径[10] PAL, 苯丙氨酸解氨酶; C4H, 肉桂酸-4-羟基化酶; 4CL, 4-香豆酸辅酶 A 连接酶; CST, 莽草酸羟基转移酶; CQT, 奎尼酸羟基转移酶; C3H, 香豆酸-3-羟基化酶; CCoAOMT, 咖啡酰辅酶 A-O-甲基转移酶; CCR, 肉桂酰辅酶 A 还原酶; CAD, 肉桂醇脱氢酶; COMT, 咖啡酸/5-羟基阿魏酸-O-甲基转移酶; F5H, 阿魏酸-5-羟基化酶; POD, 过氧化物酶; LAC, 漆酶。Fig.1 Lignin synthesis pathway PAL, Phenylalanine aminolyase; C4H, Cinnamic acid-4-hydroxylase; 4CL, 4-Coumarate coenzyme A ligase; CST, Shikimic acid hydroxyl transferase; CQT, Quinic acid hydroxyl transferase; C3H, Coumarate-3-hydroxylase; CCoAOMT, Coffee acyl coenzyme A-O-methyl transferase; CCR, Cinnamyl coenzyme A reductase; CAD, Cinnamonol dehydrogenase; COMT, Caffeic acid/5-hydroxyferulic acid-O-methyl transferase; F5H, Ferulic acid-5-hydroxylase; POD, Peroxidase; LAC, Laccase. |
2.1.1 苯丙氨酸解氨酶(PAL)
苯丙氨酸解氨酶由4个亚基组成, 4个亚基分别由多基因家族编码, 包含多种同工酶, 在植物生长发育、次生代谢以及抗逆方面起到极其重要的作用[11]。苯丙氨酸解氨酶催化苯丙氨酸脱氨基生成反式肉桂酸, 是苯丙烷代谢途径的第一个关键限速酶[12]。目前对拟南芥中PAL基因的转录调控方面的研究较为深入[13], 但由于基因表达还会受到转录后的泛素化调控和反馈调节, 导致转基因植株中PAL蛋白不能稳定表达。近年研究发现, KFB家族作为PAL蛋白的负调控因子能有效地调节PAL的稳定性和蛋白积累量[14]。迄今为止, 在梨[15]、芒果[16]、杏[17]等多种果树作物上已有PAL基因克隆以及序列分析等方面的报道, 但大多研究主要探讨不同诱导条件下PAL基因表达情况。从蜜柚汁胞中克隆得到的cmPAL基因只有一个成员[18], 分析认为PAL基因表达具有组织特异性, 表达强度随组织和器官的不同而变化。总之, PAL基因的表达在转录和翻译水平上都必然会受到多个调控因子的严格调控, 这与植物内部发育信号等条件有关, 结合转录组测序以及蛋白互作等手段, 能发现更多的调控因子, 从而调控PAL基因的稳定表达。
2.1.2 肉桂酸-4-羟基化酶(C4H)
肉桂酸-4-羟基化酶催化肉桂酸C4羟基化形成对-羟基香豆酸, 属于植物细胞色素P450的cYP73亚族, 是一种单氧化酶[19]。迄今为止, 已有拟南芥、芒果、梨、柑橘等多种植物的C4H基因被分离出来, C4H基因所编码的氨基酸序列同源性普遍较高, 是木质素生物合成途径中最保守的基因家族之一[20, 21, 22]。C4H转录丰度会影响植物中木质素、黄酮类化合物等的合成途径, C4H能够调节木质素单体G/S的比值, 其位于PAL的下游且能够调控木质素含量的变化, 因此认为C4H是更为有效的限速酶[23]。在研究琯溪蜜柚时发现, 汁胞木质素积累量与C4H表达量在果实发育的不同时期都表现出同步增加的趋势, 后期果实cmC4H表达量增加, 汁胞木质素含量增加[6]。但在奉节晚橙和奉节72-1脐橙化渣性研究中发现, 后期汁胞木质素含量差异不大, 但C4H基因表达量却不同, 可能其参与其他次生代谢物的合成[24]。
2.1.3 香豆酸-3-羟基化酶(C3H)
香豆酸-3-羟基化酶是细胞色素P450单氧加酶家族的一员, 主要催化甲酰肉桂酯类化合物。Franke等[25]通过图位克隆方法证明拟南芥中的CYP98A3是C3H基因。香豆酸-3-羟基化酶是木质素合成过程中调控碳源输送方向, 决定木质素合成类型的关键限速酶。经大量研究发现, 柑橘汁胞粒化过程中主要以G型木质素的合成为主[26], 但目前关于调控C3H的活性进而对改变植物木质素含量的研究较少, 因此, 汁胞粒化过程中C3H基因如何通过差异表达决定木质素合成的类型, 还需要更深入的研究。
2.1.4 莽草酸/奎尼酸羟基肉桂酰转移酶(HCT)
莽草酸/奎尼酸羟基肉桂酰转移酶是决定木质素最终合成H-单体和G/S-单体的关键酶之一[27]。从石榴中克隆得到PgHCT, 通过转PgHCT基因的T2代拟南芥株系进行荧光定量分析, 各组织中PgHCT基因表达量与木质素含量呈显著正相关[28]。在琯溪蜜柚中克隆得到cmHCT, 经亚细胞定位发现cmHCT主要定位于质膜上, 并结合HCT的表达特征, 认为汁胞木质素的合成以G型木质素为主要途径[18]。C3H和HCT处于木质素生物合成的上游, 在木质素生物合成过程中有相似的功能。形成H-木质素和G/S-木质素单体关键取决于C3H和HCT的活性[29]。
2.1.5 4-香豆酸辅酶A连接酶(4CL)
4-香豆酸辅酶A连接酶是木质素合成途径上游的一个关键酶, 可将羟基肉桂酸催化生成相应的羟基肉桂酸CoA酯, 进而参与后续的木质素、黄酮素类等的合成[30]。4CL通常以基因家族的形式存在于植物中, 当植物遭受非生物胁迫等逆境时, 不同亚型的4CL会参与不同的代谢。在枇杷木质化的过程中, 初步确定Ej4CL1在枇杷果实木质素合成过程中发挥了重要作用, 并采用烟草过量表达体系验证了Ej4CL1基因功能, 过量表达Ej4CL1能够显著增加烟草叶片木质素积累并减少单宁积累[31]。通过生物信息学分析, 从克里曼丁柑橘基因组数据库筛选鉴定出3个cit4CL基因, 对其在北京柠檬不同组织和果实发育过程中的表达进行了分析, 通过聚类分析将Cit4CL2和Cit4CL3基因聚到了主要参与木质素生物合成的ClassⅠ 一类[32]。
2.2.1 肉桂酰辅酶A还原酶(CCR)
肉桂酰辅酶A还原酶催化羟基肉桂酸CoA酯氧化还原为相应醛类物质, 是决定木质素单体合成类型的关键酶之一。迄今为止, 已有21种植物的CCR基因被克隆并进行研究分析[33, 34, 35]。植物中主要存在两种类型的CCR, 其中CCR1主要参与植物木质素的合成, 而CCR2主要参与植物逆境生理[36]。进一步研究发现, 植物的不同组织中CCR基因存在组织差异性表达, 但组织中木质素含量与CCR的表达量基本一致[37]。在研究授粉对琯溪蜜柚果心汁胞木质素合成影响时发现, 木质素含量的变化与果心汁胞CCR1表达量呈正相关, 而CCR2的表达量与汁胞木质素含量呈负相关[6]。
2.2.2 阿魏酸-5-羟化酶(F5H)
阿魏酸-5-羟化酶是木质素生物合成中关键酶之一, 对阿魏酸、松柏醛、松伯醇都具有催化作用, 生成相应的5-羟基阿魏酸、5-羟基松柏醛和5-羟基松柏醇, 是S木质素单体合成的前提[38]。F5H是一种P450依赖的单加氧酶, 通过生物信息学分析推测其是位于内质网上的具有跨膜结构域的亲水蛋白[39]。近年来, 已从梨[40]、石榴[41]等果树中克隆出了F5H基因。研究发现, PbF5H是调控砀山酥梨果实S木质素合成的关键基因之一, 影响木质素单体G/S比值, 进而影响石细胞含量[40]。通过对桑树F5H基因生物信息学分析发现, F5H上游启动子中无保守的AC元件, 该基因表达不受MYB转录因子的调控, 而是直接受NAC结构域的转录因子的调控[42]。
2.2.3 咖啡酸邻位甲基转化酶(COMT)
咖啡酸邻位甲基转化酶在木质素生物合成过程中主要参与甲基化反应, 是决定木质素单体的类型以及组成比例的关键酶之一[43]。研究发现, COMT是多基因家族, 在木质素生物合成过程中, 参与了多条甲基化反应[44]。COMT可以单独或同时催化3'和5'位置的醛或醇, 能够分别将咖啡醛、咖啡醇转化成松柏醛、松柏醇[45], 将5-羟基松柏醛和5-羟基松柏醛分别转化成芥子醛和芥子醇[46]。研究发现, 在烟草和杨树中抑制COMT基因的表达, S/G比值下降, 且S型木质素含量显著下降, G型木质素含量增加但变化不显著[47]。但在琯溪蜜柚粒化汁胞中并未获得COMT基因片段, 根据前人研究柑橘中主要以G型木质素存在, 因此推测粒化汁胞中可能COMT表达量较少, 甚至不表达, 但具体原因尚无相关研究。
2.2.4 咖啡酰辅酶A-3-O-甲基转移酶(CCoAOMT)
咖啡酰辅酶A-3-O-甲基转移酶在木质素生物合成中主要催化羟基辅酶A酯的甲基化反应, 将咖啡酰辅酶A转化成阿魏酰辅酶A[48]。咖啡酰辅酶 A-3-O-甲基转移酶是多基因家族, 从现有的研究可以发现, 甲基化反应的关键酶在决定木质素单体结构和组成上有显著效果, 在转基因苜蓿中, CCoAOMT被抑制造成G木质素下降50%, 但对S木质素没有影响[49]。研究者运用反义RNA等技术抑制拟南芥中CCoAOMT的活性, 不但能降低木质素的含量, 还能使其单元组成发生改变, 从而降低G/S值[50]。由于其多以基因家族形式出现, 对于其研究有一定的困难。迄今为止, 柑橘类CCoAOMT基因功能未见报道, 但随着基因编辑技术Cas9/gRNA的不断完善, 为进一步深入研究柑橘类CCoAOMT基因功能奠定了基础。
2.2.5 肉桂醇脱氢酶(CAD)
肉桂醇脱氢酶主要将三种肉桂醛还原生成相应的3种肉桂醇(香豆醇、芥子醇、松柏醇)[51]。由于同工酶的存在, 该酶在不同植物及相同植物不同组织中的催化功能有较大的差异。在拟南芥中分离出的AtCAD4和AtCAD5在高活性水平的条件下对G/S-木质素单体的合成有促进作用[52]。研究发现, 在烟草中同时下调CCR和CAD基因的表达, 植株生长发育正常, 但木质素含量显著下降, 推测两者之间可能存在互作关系, 但具体机制还要进一步研究[53]。红肉脐橙在干旱/低温胁迫下, CAD表达量上升, 汁胞细胞壁合成大量的木质素[2]。在琯溪蜜柚中克隆得到2个CAD基因, 序列差异不大, 通过亚细胞定位, 经PSORT分析cmCAD1可能存在于内质网上, 推测与汁胞粒化及前期细胞中高尔基体增加相关, 但需要进一步验证该基因的功能[18]。
2.2.6 过氧化物酶(POD)
过氧化物酶是由单一肽链和卟啉构成的血红素蛋白, 拥有庞大的基因家族, 广泛存在于植物体内的不同组织, 参与活性氧代谢、木质素代谢等途径[54]。POD作为木质素合成途径的关键酶, 经过苯丙烷代谢合成的木质素单体, 通过该酶催化作用发生脱氢聚合反应形成木质素[55]。按不同的催化底物POD可分为:谷胱甘肽POD(GSH-PX)、愈创木酚POD(PPOD)和抗坏血酸POD(APX)[56]。以往的研究主要集中在木质素与POD的关系上, 如南丰蜜桔汁胞木质素含量、芥子醇含量、松伯醇含量与POD活性呈极显著正相关[57]。研究发现, 砀山酥梨果实木质素代谢中主要以PPOD类型为主进而参与木质素单体的聚合, 促进梨石细胞的形成[58]。柚汁胞中POD主要以可溶性和离子结合状态存在, 粒化汁胞中的POD为离子结合态的碱性POD, 但POD参与粒化汁胞木质素形成的分子机制还有待进一步的探索[59]。
2.2.7 漆酶(LAC)
漆酶属于铜蓝蛋白家族, 含有500个左右的氨基酸, 以氧气为电子受体, 具有丰富的催化底物。随着研究的深入, 漆酶已被证明对植物次生细胞壁的形成起到关键作用, 是木质素单体最终聚合的关键酶[60]。近年来, 相继在棉花[61]、高粱[62]、水稻[63]中验证了漆酶基因在木质素合成过程中起到关键作用。在柑橘上研究发现, 枳壳受到过量硼胁迫处理时, miR397可通过调节LAC7的活性促进木质素的合成, 从而提高植株对硼毒害的耐受性[64]。通过生物信息学分析, 甜橙基因组鉴定出24个漆酶基因(CsLAC1~CsLAC24), 在大多数CsLAC中, 有4个保守的特征序列和3个典型的特征序列, 且观察到Cu-氧化酶结构域。系统聚类分析表明, 甜橙漆酶基因组CsLACs可分为7组, 这表明其存在着不同的功能和作用, 为进一步研究漆酶在柑橘中的功能提供了有价值的线索[65]。
在木质素生物合成过程中不仅需要多种酶基因的协调参与, 同时也受到严格的转录调控。研究发现, NAC转录因子和一些MYB转录因子在木质素合成中有重要调控作用[66]。如图2所示, 在拟南芥中, SND1及其同源蛋白NST1、NST2、VND6、VND7位于次生壁合成网络的上游, MYB46、MYB83是它们的直接靶蛋白[67]。MYB46、MYB83作为直接靶蛋白受SND1及其同源蛋白 NST1、NST2、VND6、VND7的直接调控, MYB58、MYB63以及MYB85作为特异转录因子受MYB46、MYB83的调控, 形成相应的转录网络对木质素的合成进行调控[68]。后续通过某些MYB类转录因子与苯丙烷代谢途径中与木质素合成相关基因上游启动子的AC元件结合, 对木质素生物合成进行调控[69]。在甜橙汁胞粒化的研究中分离并鉴定了CsMYB330和CsMYB308转录因子, 发现基因的表达与木质素含量呈正相关, 过表达该基因可以诱导木质素的积累, 这表明了这两种转录因子可能是柑橘汁胞木质素合成的转录因子[70]。进一步鉴定并分离了转录因子CsMYB85, 发现在汁胞粒化过程中, CsMYB85表达水平显著增加。CsMYB85结合CsMYB330启动子, 调控其表达, 瞬时表达试验表明, 随着CsMYB85的过量表达, 汁胞中4CL1的表达量与木质素的含量变化趋势一致, 推测该转录因子通过调控4CL基因转录表达从而调控木质素的生物合成[71]。除NAC和MYB外, NtLIMl、ACBF和bHLH类转录因子也通过其他途径参与调控木质素的生物合成[72]。
目前在柑橘粒化过程中对木质素代谢研究存在的问题主要有:(1)更多的研究是针对木质素合成途径中单个基因的功能研究, 该基因表达量的上调和下调与木质素含量之间的关系仍有待进一步探讨; (2)柑橘粒化汁胞中与木质素合成相关基因的功能尚不能明确; (3)转录因子调控木质素生物合成, 形成柑橘汁胞粒化的机制尚不明确; (4)现有的研究多以实验室试验为主, 大田实验进行较少, 成果转化率较低。结合当前研究存在的问题, 今后对柑橘汁胞粒化过程中木质素生物合成的研究应集中在以下几点:(1)利用转录组学、蛋白组学、代谢组学等现代生物技术发掘更多与木质素合成的关键基因, 确定关键基因之间的关系, 基于多组学的网络构建共同完善木质素生物合成调控网络; (2)在柑橘粒化汁胞中克隆出相关调控木质素合成基因后, 通过转基因技术转入模式植物中进行功能验证, 并进一步构建柑橘自身转基因体系进行功能验证; (3)结合目前已经完成测序的甜橙基因组, 利用RNA干扰技术发掘更多与木质素生物合成相关的转录因子; (4)广泛收集柑橘种质资源, 针对不同柑橘品种汁胞粒化进行对比分析, 结合大田试验, 探索能解决柑橘汁胞粒化的方法, 使研究成果有效转化到实际生产之中。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
|
| [54] |
|
| [55] |
|
| [56] |
|
| [57] |
|
| [58] |
|
| [59] |
|
| [60] |
|
| [61] |
|
| [62] |
|
| [63] |
|
| [64] |
|
| [65] |
|
| [66] |
|
| [67] |
|
| [68] |
|
| [69] |
|
| [70] |
|
| [71] |
|
| [72] |
|


