作者简介:王伟科(1981—),男,浙江宁海人,硕士,高级农艺师,研究方向为食药用菌育种与栽培技术。E-mail: akeok@126.com
为保护开发野生桑树桑黄种质资源,对17个不同来源的野生桑树桑黄菌株开展种内遗传多样性分析,利用简单重复序列间扩增(ISSR)分子标记技术对桑黄菌株DNA进行扩增,分析扩增条带,利用NTSYS软件构建亲缘关系UPGMA聚类图。结果表明:16条ISSR引物中,有10条ISSR引物多态性丰富,条带清晰;10条引物共检测到904个位点,其中,多态性位点717个,多态性百分比79.3%。在DNA指纹图谱中,引物P5、P812扩增条带多态性最高。NTSYS-PC2.10e软件分析表明,17个桑树桑黄遗传相似系数为0.57~0.99。UPGMA聚类分析结果显示,在遗传相似系数(GS)约0.65处,可将17个桑树桑黄划分为2大类群: S4,S23,S26为一大类群,其余为一大类群。综上可知,桑树桑黄种质资源具有丰富的遗传多样性,ISSR分子标记可有效区分不同桑树桑黄菌株。
In order to provide basis for protection and development of germplasm resources of wild Sanghuangporus sanghuang, the genetic diversity of 17 wild Sanghuangporus sanghuang strains was investigated in this study. The amplified products of 17 wild Sanghuangporus sanghuang strains DNA based on ISSR makers technique were used to analyze the genetic diversity of Sanghuangporus sanghuang. Cluster dendrogram of different samples were established based on the unweighted pair-group method with arithmetic mean (UPGMA) by NTSYS-pc software. The results showed that bands amplified by 10 ISSR primers among the 16 ISSR primers were clear and polymorphisms rich. 10 ISSR primers generated 904 loci, of which 717 loci were polymorphic. The percentage of polymorphisms was 79.3%. Bands amplified by ISSR-primers P5 and P812 had the highest polymorphisms in DNA fingerprint. The genetic similarity coefficients of 17 Sanghuangporus sanghuang strains ranged from 0.57 to 0.99. UPGMA cluster analysis showed that 17 Sanghuangporus sanghuang strains were divided into 2 groups at the level of genetic similarity coefficient (GS) about 0.65 in the cluster dendrogram. S4, S23, S26 were grouped together and the other 14 strains were in one group. Results of ISSR analysis revealed that germplasm resources of Sanghuangporus sanghuang had plentiful genetic diversity. ISSR method was efficient for distinguishing different strains of Sanghuangporus sanghuang.
桑黄隶属于真菌界(Fungi)、担子菌门(Basidiomycota)、层菌纲(Hymenomycetes)、非褶菌目(Aphyllophorales)、锈革孔菌科(Hymenochaetaceae)、桑黄属(Sanghuangporus), 是一种珍贵的药用真菌, 因寄生于桑树而得名, 别称桑臣、桑耳[1]。现代科学研究证实, 桑黄具有抗肿瘤、降血糖、护肝、抗炎、免疫调节、抗氧化等药理作用, 具有极高的开发利用价值[2]。桑黄属种质鉴定、新品种选育及高效栽培技术一直是科研人员研究关注的重点[3]。在前期研究中, 利用ITS (internal transcribed spacer)序列分析技术, 对从杭州、丽水及安徽金寨等地采集的22个桑黄样品进行了鉴定, 成功区分出了杨树桑黄(Fuscoporia gilva)及丁香桑黄(Inonotus baumii), 并得到了17份桑树桑黄(Sanghuangporus sanghuang)样品(样品采集于不同地区的野外桑树树干)。鉴于ITS在种内间隔区上表现的差异较小, 不适宜种内菌株的进一步区分, 需利用分子标记技术进一步研究上述试验中鉴定得到的桑树桑黄种内遗传多样性。
目前, ISSR(inter simple sequence repeat)、AFLP(amplified fragment length polymorphism)、RFLP(restriction fragment length polymorphism), RAPD(random amplified polymorphic DNA)等分子标记技术已广泛运用于药用植物及食用大型真菌种类遗传多样性鉴定[4]。ISSR分子标记技术以其高效、快速、稳定、可靠等诸多优点在物种遗传多样性鉴定、系统进化等多个领域得到广泛运用。大量研究表明, ISSR分子标记技术可以用于种群间或种群内的遗传多样性分析[5]。在本研究中, 我们拟采用ISSR分子标记技术对前期研究中获得的17个桑树桑黄样品的遗传多样性进行分析, 以期明确桑树桑黄种内样品之间的亲缘关系。同时, 为规范桑树桑黄引种栽培、加强资源保护和品种选育打下基础。
测试菌种(株)来自我国各主要桑黄真菌保藏单位, 经前期ITS序列鉴定为桑树桑黄样品(表1)。
| 表1 桑树桑黄真菌测试菌株来源 Table 1 Strains used for Sanghuangporus sanghuang genetic diversity study |
从培养5~7 d 的PDA培养基平板上挑取2 mm × 2 mm的菌丝块, 接种于PDA液体培养基, 25~27 ℃浅层静置培养15~20 d。然后收集菌丝体, 用无菌水漂洗2次, 灭菌滤纸吸干水分, 用于提取基因组DNA。
取0.1 g菌丝体, 液氮研磨后加入2% CTAB, 用于基因组DNA的提取[6]。用0.8%琼脂糖凝胶电泳和NanoDrop 2000 C超微量分光光度计(Thermo)检测所提DNA浓度和纯度。DNA样品稀释至40 ng· μ L-1, 用于ISSR扩增。
ISSR引物参考哥伦比亚大学公布的序列及Wolfe等[7]的文献, 从中筛选出16条合适引物, 引物由北京擎科生物技术有限公司合成。引物序列见表2。
| 表2 ISSR引物参考序列 Table 2 Sequences of tested ISSR primers |
ISSR扩增体系根据各自引物退火温度及Mg2+浓度进行优化, 优化后体系含10× PCR buffer 2 μ L、25 mmol· L-1 MgCl2 1.2 μ L、10 mmol· L-1 dNTPs 0.5 μ L、10 μ mol· L-1 ISSR引物各1 μ L、80 ng基因组模板DNA及1 U pfu DNA聚合酶, 反应总体积20 μ L。
PCR扩增程序为: 94 ℃ 3 min; 94 ℃ 20 s, 46~52 ℃ 90 s, 、72 ℃ 30 s, 35个循环; 72 ℃ 7 min。其中, 每个引物的退火温度优先参考值为Tm值下调2 ℃。
取目标产物10 μ L, 加入6× 上样缓冲液1.5 μ L, 混匀后于1%琼脂糖凝胶上电泳分离。电泳缓冲液采用TAE, 稳压电泳, 电压120 V, 40 min后停止电泳并拍照记录。
从凝胶成像系统得到的电泳谱带中, 选取清晰可辨的电泳条带, 以“ 1” 和“ 0” 记录条带的有或者无, 在相同片段位置上存在扩增带时, 记录为“ 1” , 不存在时记录为“ 0” , 将图形数据转换成数字数据。并运用NTSYS-pc2.0软件, 根据SM相似系数法(coefficient)计算样品间的遗传相似性, 用UPGMA法进行聚类分析。
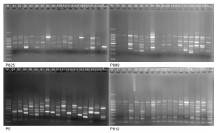
用来扩增桑树桑黄的16条ISSR引物中, 有10条获得的ISSR带型较好, 各引物扩增获得的条带片段大小范围较大, 为200~3 000 bp(表3)。这10条引物共扩增获得904条DNA条带, 其中, 多态性条带717条。引物P816、P817、P825、P828扩增的多态性比率最高, 为100%; 引物P810扩增的多态性比率最低, 为55.7%(表3)。
| 表3 10条ISSR引物扩增结果 Table 3 Amplification results of 10 different ISSR primers |
在桑树桑黄ISSR扩增DNA指纹图谱中(图1), 引物P825、P889分辨力最高, 对桑树桑黄基因组DNA扩增效果最好, 引物P5、P812扩增条带多态性最高, 表明用该ISSR引物建立的指纹图谱用来分析桑树桑黄遗传多样性较好。
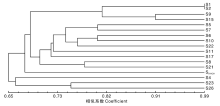
聚类分析结果表明, 在遗传相似系数约0.65处, 17个菌株可划分为2大类群:S4、S23、S26为一大类群; 其余14个菌株为一大类群, 在这一大类群中, 在遗传相似系数为0.73处, S11、S17、SKHCH各单独聚为一类, S8, S21聚为一类, S5、S6、S7、S10、S22聚为一类, S1、S2、S9、S15聚为一类(图2)。
17个桑树桑黄的遗传相似系数(表4)结果表表明, 17个菌株的遗传相似系数为0.57~0.99, 平均遗传相似系数为0.71。其中, S2与S26之间的遗传相似系数最低, 为0.58, 可见S2与S26之间的亲缘关系最远。S1与S2之间的遗传相似系数最高, 为0.99, 说明S1与S2之间的亲缘关系最近。
| 表4 17个桑树桑黄菌株的遗传相似系数 Table 4 Genetic similarity coefficient of 17 strains of Sanghuangporus sanghuang |
用表型特征和生理生化特征等传统方法来鉴定野生桑树桑黄子实体需对菌株开展出菇试验, 但桑黄生长缓慢, 很难在短期内获得子实体, 且其子实体表型易受外部环境影响, 这些因素使得桑黄鉴定难度增加。近年来, 迅速发展的DNA分子标记技术因其具有鉴定快速、不受外界环境干扰、直接反映样品遗传本质等优点, 被越来越多地用在食药用菌种间、种内的遗传多样性分析中。
本研究利用ISSR-PCR技术对经ITS分子标记鉴定后获得的17个桑树桑黄种内菌株的遗传多样性进行分析, 有10条引物扩增产物多态性丰富, 条带清晰, 获得DNA条带904条, 其中多态性条带717条。这表明桑树桑黄种内菌株的遗传多样性比较丰富。用UPGMA进行聚类分析表明, 在遗传相似系数约0.65处, 17份样品可划分为2大类群, S4、S23、S26为一大类群, 其余为一大类群。这表明ISSR分子标记能有效地揭示桑树桑黄种质资源间丰富的多态性和遗传多样性。
基于ISSR分子标记技术对桑树桑黄种内菌株的遗传多样性及亲缘关系进行分析, 不仅能对桑黄开展系统进化生物学研究, 也能为我国桑黄资源开发利用、品种选育、栽培技术研究提供理论依据[8]。同时, 对桑黄菌株进行分类鉴定不能只依靠单一分子标记技术, 需进一步结合菌丝形态、子实体特征、生理生化特性等开展系统的研究和分析, 从而明确桑黄菌株之间的分类定位[9, 10]。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|