作者简介:秦玲(1994—),女,四川遂宁人,硕士,研究方向为果树生理生态。E-mail:875332955@qq.com
从金冠基因组数据库中,利用BLAST网站在苹果中鉴定得到8个编码多胺氧化酶(polyamine oxidase,PAO)的家族基因(命名为 MdPAO1~8),对其进行理化性质、系统进化、蛋白结构、基因结构和启动子元件分析。结果表明,MdPAO蛋白的分子量为54.053~64.813 ku,等电点介于5.16~6.02。根据进化树分析,MdPAO被分为3个亚家族(Ⅰ、Ⅲ、Ⅳ),且同一亚家族的成员具有相似的蛋白结构、基因结构和蛋白保守基序分布。利用GEO数据库检测出8个 MdPAO在不同组织器官中的表达具有明显差异。以苹果主栽品种长富2号为材料,利用实时荧光定量PCR检测分析得出8个 MdPAO在不同组织中的表达情况,结果表明, MdPAO3、 MdPAO4、 MdPAO5/6和 MdPAO8在花中有较高的表达水平; MdPAO1/2和 MdPAO7在茎和果中有较高的表达水平。外源亚精胺处理后发现,除 MdPAO3以外,其余 MdPAO的转录水平显著提高,表明 MdPAO在PA代谢途径中具有重要作用。
From the genomic database of Golden Delicious, eight family genes encoding polyamine oxidase (PAO) (named as MdPAO1-8) were identified in apples using the BLAST website for physicochemical properties, phylogenetic, protein structure, analysis of gene structure and promoter elements. The results showed that the molecular weight of MdPAO protein ranged from 54.053 to 64.813 ku, and the isoelectric point ranged from 5.16 to 6.02. According to the phylogenetic tree analysis, MdPAO is divided into three subfamilies (Ⅰ,Ⅲ, Ⅳ), and members of the same subfamily share similar protein structure, gene structure and protein conserved motif distribution. The expression of 8 MdPAO genes in different tissues and organs was significantly different using the GEO database. The expression of 8 MdPAO genes in different tissues was determined by real-time quantitative PCR using Nagafu No.2. The results showed that MdPAO3, MdPAO4, MdPAO5/6 and MdPAO8 were highly espressed in flowers. MdPAO1/2 and MdPAO7 had higher expression levels in stems and fruits. Exogenous spermidine treatment showed that the transcriptional level of MdPAO significantly increased except MdPAO3, indicating that MdPAO plays an important role in the PA metabolic pathway.
多胺(polyamine, PA)是普遍存在的低分子量有机阳离子, 包括腐胺(putrescine, Put)、亚精胺(spermidine, Spd)、精胺(spermine, Spm)和热精胺(thermospermine, T-spm)[1]。大量研究表明, PA在植物生长和发育过程中是必不可少的, 包括花芽分化、开花调节、生殖器官发育、性别分化、果实生长和衰老[2]。据报道, 两种类型的胺氧化酶, 包括含铜胺氧化酶(copper-containing amine oxidases, CuAOs)和FAD依赖性多胺氧化酶(FAD-dependent polyamine oxidases, PAOs)参与PA的分解代谢[3]。植物PAO负责末端分解代谢或PA的反向转化, 从而产生相应的二氨基丙烷和氨基醛, 并释放过氧化氢(H2O2)[4, 5]。
目前, PAO基因家族成员的鉴定已在拟南芥[6]、水稻[7]、大麦[8]、玉米[9]、烟草[10]、杨树[11]、甜橙[12]、陆地棉[13]、番茄[14]等植物中有过报道, 且在不同植物中PAO家族成员的数量表现出差异性。例如, 拟南芥含有5个PAO基因(AtPAO1-AtPAO5), 甜橙含有6个PAO基因(CsPAO1-CsPAO6), 水稻含有7个PAO基因(OsPAO1-OsPAO7), 陆地棉含有12个PAO基因(GhPAO1-GhPAO12)。到目前为止, 拟南芥、水稻和番茄等模式植物中PAO基因家族得到广泛关注和研究, 但木本植物中关于PAO家族成员的研究还比较少, 仅在苹果中报道过两种编码PAO的cDNA(MdPAO1和MdPAO2a)[15], 但没有对苹果PAO基因家族进行系统完整的生物学分析。
根据研究表明, 植物中的PAOs通常分为4个亚家族[16, 17]。亚家族Ⅰ 的成员参与Spm的反向转化, 位于细胞质中; 亚家族Ⅲ 和Ⅳ 的成员在Spm逆转化途径中也起到降解PAs的作用, 前者位于细胞质中[5, 18], 后者位于过氧化物酶体中[7]。而亚家族Ⅱ 的成员负责PAs的最终分解代谢, 位于外质体[19]或液泡中[20]。
PA参与对生物和非生物胁迫的防御反应[1]。研究表明, 利用转基因方法和外源PA可增强植物对盐胁迫的耐受性[21]。植物PAO负责PA末端分解代谢或反向转化, 这些氧化途径可能影响PA稳态并调节激素信号, 从而改变植物对胁迫的耐受性[22]。在三个主要的PA中, Spd与植物的胁迫耐受性联系更密切[23]。本实验通过外源Spd处理, 分析在植物生理生化进程中PAO编码基因在转录水平上的相对表达量, 研究苹果PAO基因在PA代谢中的可能作用, 为进一步研究PAO基因在胁迫中的功能提供依据。另外, 有关苹果PAO基因家族成员系统的生物信息学分析也鲜见报道。本研究根据已有的金冠基因组数据库和相关的生物信息学方法, 系统鉴定苹果PAO 家族成员, 分析其进化关系、基因结构和基因组织表达等, 研究不同组织(花、茎、叶和果实)中其表达量的变化规律, 为深入研究苹果PAO基因的功能提供理论基础。
在TAIR网站(http://www.arabidopsis.org/)根据文献报道的基因登录号下载拟南芥5个AtPAO的基因序列和蛋白序列[6]。在GDR苹果全基因组数据库Blastp工具中初步比对获得苹果中候选PAO基因登录号, 在金冠参考基因组数据库中下载其相应的蛋白序列、基因序列、基因定位及基因注释信息。在NCBI (https://www.ncbi.nlm.nih.gov/) 中根据文献报道的基因登录号下载多个来源于单子叶和双子叶植物的PAO蛋白序列, 构建系统进化树。在GEO数据库(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE42873) 中得到金冠以及不同苹果杂交种中不同器官的PAO基因表达数据(GSE42873)。
实验于2019年在陕西省西北农林科技大学宝鸡千阳苹果试验示范站(107° 13'E, 34° 65'N) 进行。材料选自七年生长富2号/M26/八棱海棠, 植株行距为1.3 m × 4.0 m, 果园采用统一的矮化密植高纺锤形栽培模式, 树势中庸。选取负载量和长势基本一致的植株18株, 分成6个小区, 每个小区3棵树。根据曹尚银等[24]研究, 随机选择3个小区在盛花期后25 d(2019年5月5日)和盛花期后30 d(2019年5月10日)对整树喷施1× 10-5 mol· L-1 Spd两次, 其他3个小区喷施清水作为对照。所有处理采用手动喷雾器, 喷施的湿润程度为叶片和茎表面充分湿润但无液滴凝聚下落。
在对照植株中, 于盛花期采集短枝顶花, 盛花期后30 d采集幼果(直径2 cm左右)、短枝顶芽毗邻叶和短枝茎(直径2~3 mm)[25], 用于组织特异性分析。对所有植株, 于盛花期后30 d开始, 分别在生理分化前、中、后期采集长富2号的叶片(短枝顶芽毗邻叶), 共采集3次(盛花期后30、50、70 d)[26], 对PAO基因进行表达分析。所有采集材料(茎, 花, 叶, 幼果包括果肉和果皮) 均用液氮冷冻处理, -80 ℃保存, 用于RNA提取。
以获得的5个AtPAO基因的蛋白序列作为检索依据, 在苹果基因组数据库GDR中进行Blastp比对(https://www.rosaceae.org/blast/protein/protein), 选择估计值小于e-5的苹果基因登录号[27], 去掉重复的基因。依据在金冠参考基因组数据库中下载的相应蛋白序列, 利用Pfam数据库(https://pfam.xfam.org/search/sequence)确定只含有氨基氧化酶结构域(PF01593)的基因, 最终鉴定出苹果PAO基因。根据染色体的位置情况对苹果中的PAO依次命名。利用ExPaSy在线网站(https://web.expasy.org/protparam/) 分析MdPAO蛋白的理化性质[28]。
利用DNAMAN软件, 采用系统默认参数值, 对苹果MdPAO和玉米ZmPAO1蛋白进行多重序列比对[29]。
通过文献报道的基因登录号在NCBI中下载获得多个单子叶和双子叶植物的PAO蛋白序列[12, 14], 利用MEGE6.0软件构建多物种系统进化树, 来探讨PAO蛋白的进化关系, 并分析得出苹果MdPAO所属亚家族。方法采用邻接法(neighbor-joining, NJ), 校验参数选择Bootstrap重复1 000次, 模式采用p-diastance model, 缺口设置选择partial deletion[30]。
SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)在线工具对MdPAO蛋白的二级结构进行预测[31], 包括分析其α -螺旋、β -折叠和无卷曲情况。并在线利用PHYRE serve v2. 0(http://www.sbg.bio.ic.ac.uk/phyre2htmlpage.cgi?id=index)对MdPAO蛋白进行三级结构的预测[32]。
利用在线网站WoLF PSORT (https://wolfpsort.hgc.jp/)预测MdPAO蛋白的亚细胞定位[33]。
MdPAO基因的外显子-内含子结构示意图利用在线工具Gene Structure Display Serve 2.0(GSDS)(http://gsds.cbi.pku.edu.cn/)获得[34]。MdPAO蛋白的保守基序分析利用MEME (http://meme-suite.org/tools/meme)在线网站完成, 设置最大保守基序数量为10, 其余参数为默认设置值[35]。从金冠基因组数据库中选取MdPAO基因转录起始位置(ATG)上游1 500 bp的序列作为启动子区, 利用在线数据库PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/), 对其启动子的顺式作用元件进行分析[36, 37]。
采用改良CTAB法[38]提取不同组织部位的总RNA, 取完整性良好的总RNA经RNase-Free DNase处理之后, 去除含有的苹果DNA。使用TaKaRa PrimeScriptTM RT reagent试剂盒, 按照产品说明书合成cDNA。
利用实时荧光定量PCR分析MdPAO基因在不同组织与亚精胺处理中的表达情况。采用软件Primer premier 5.0设计MdPAO特异性引物(表1), 由于MdPAO2和MdPAO3拥有相似的编码序列, 不能设计特异性引物区分, 所以它们共用一对引物; MdPAO5和MdPAO6也拥有相似的编码序列, 因此共用一对引物。内参基因选用苹果EF-1α (GenBank 登录号DQ341381), 其特异性引物为:EF-1α -F, 5'-ATTCAAGTATGCCTGGGTGC-3'; EF-1α -R, 5'-CAGTCAGCCTGTGATGTTCC-3'[39]。以上述cDNA为模板, 参照试剂盒TaKaRa SYBR ® Premix EX TaqTM Ⅱ 说明书在QuantStudio5定量仪上进行qRT-PCR反应。反应体系为10 μ L(其中SYBR ® Premix Ex TaqTM Ⅱ 2× 为5 μ L, 上、下游引物各0.5 μ L, 引物浓度为10 μ mol· L-1, cDNA为1 μ L, ddH2O为3 μ L)。反应程序为95 ℃预变性3 min, 94 ℃变性15 s, 60 ℃退火20 s, 72 ℃延伸20 s, 40次循环[40], 均设置3次重复, 用2-Δ Δ Ct方法计算基因的相对表达量[41]。
| 表1 MdPAO定量引物序列 Table 1 Primer sequenceces of MdPAO genes |
利用GEO在线数据库对MdPAO基因的组织特异性进行分析。从GEO数据库(登录号为GSE42873)下载金冠以及不同苹果杂交种不同器官的基因表达数据, 从中提取MdPAO相关基因的表达量[42], 并利用HEML1.0软件作图。
所有数据(表示为3次重复的平均值)在IBM SPSS v19中进行单因素方差分析(ANOVA), 采用Duncan的多范围检验(P< 0.05)进行显著性分析。相关图表制作使用Excel 2010。
通过生物信息学手段, 以拟南芥AtPAO的基因家族为检索靶标, 从苹果基因组中获得了8个PAO基因, 根据其染色体的位置, 分别命名为MdPAO1-MdPAO8(表2)。
| 表2 MdPAO基因家族 Table 2 MdPAO gene family |
通过在线网站ExPaSy对MdPAO基因和MdPAO蛋白的理化性质进行了分析, 结果如表2所示。MdPAO基因的CDS序列长度较长, 均为1 400 bp以上, 其中MdPAO6的CDS序列最长; MdPAO蛋白包含490(MdPAO4)~582(MdPAO6)个氨基酸, 分子量在54.053(MdPAO4)~64.813(MdPAO6)ku, 等电点介于5.16(MdPAO3)~6.02(MdPAO5), 均略小于7, 呈酸性, 是酸性蛋白。进一步发现, 所有MdPAO蛋白的不稳定系数均小于40, 证明都是稳定蛋白。MdPAO5平均亲水性系数大于0, 其余MdPAO蛋白都小于0, 即MdPAO5的疏水性最强, 其余MdPAO蛋白均有很好的亲水性, 且亲水程度不同。另外, 这8个MdPAO基因分别定位在2号、9号、15号和17号染色体上, 其中2号和9号染色体都富集了3个MdPAO基因, 15号和17号染色体上均仅存在1个MdPAO基因。这些结果可以为研究MdPAO蛋白的功能提供借鉴。
为了研究MdPAO蛋白序列的特征, 用DNAMAN软件对MdPAO和ZmPAO1进行多重蛋白序列的比对。结果显示(图1), MdPAO成员与ZmPAO1表现出不同的序列同源性, MdPAO成员之间也表现出差异性。该分析预测了MdPAO4、MdPAO6和MdPAO8中过氧化物酶体靶向序列的存在。
 | 图1 MdPAO和ZmPAO1氨基酸序列的比对 灰色背景着色为相同和相似的氨基酸残基; 序列上方的黑线表示人类MAO-B和ZmPAO1的FAD结合区域[6, 43]; 黑色方框表示过氧化物酶体靶向信号[23, 44]。Fig.1 Alignment of the amino acid sequence of MdPAO and ZmPAO1 Gray background colored were the identical and similar amino acid residues; Black lines above the residue indicated the FAD binding region of human MAO-B and ZmPAO1[6, 43]; Black boxes represented peroxisome targeting signals[23, 44]. |
为了研究不同物种PAO的系统进化关系, 利用MAGE软件, 采用NJ法构建了一个包含36条来自不同物种的系统发育树(图2), 其中苹果8条, 拟南芥5条, 甜橙6条, 水稻7条, 番茄7条, 大麦2条, 玉米1条。结果显示, 苹果MdPAO与番茄、拟南芥和甜橙的PAO亲缘关系较近, 与水稻、玉米和大麦亲缘关系较远, 原因可能是苹果与甜橙和番茄等双子叶植物分离进化时间更近。
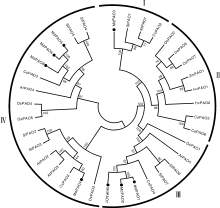 | 图2 多个物种中PAO蛋白系统进化树 蛋白序列由文献报道的基因登录号在NCBI查询获得[12, 14]。Fig.2 Phylogenetic tree of PAO proteins of different varieties The protein sequence was obtained from the NCBI query by the gene landing number reported in the literature[12, 14]. |
结果表明, PAO家族分为4个亚家族, 且每个亚家族内, 基因数分布不均匀, 分别包含4、8、8和16个。其中MdPAO3属于亚家族Ⅰ ; 而没有苹果MdPAO属于亚家族Ⅱ ; 3个苹果MdPAO(MdPAO1、MdPAO2和MdPAO7)属于亚家族Ⅲ ; 4个苹果MdPAO(MdPAO4、MdPAO5、MdPAO6和MdPAO8)属于亚家族Ⅳ 。
利用在线网站ExPaSy对MdPAO蛋白进行一级结构的预测, 发现同一亚家族成员氨基酸的组成与含量相差较小; 不同亚家族成员之间差异较大。利用在线网址SOPMA对MdPAO蛋白二级结构预测的结果显示(表3), MdPAO蛋白的二级结构由α -螺旋、β -转角、延伸链和无规则卷曲组成, 其中主要组成部分是α -螺旋和无规则卷曲, 延伸链次之, β -转角占很小的一部分(6%左右)。二级结构进一步折叠形成三级结构(图3), 利用PHYRE serve v2.0预测MdPAO蛋白的三级结构, 同源性越高, 其相似度越高。
| 表3 MdPAO蛋白一级结构、二级结构及亚细胞定位预测 Table 3 Primary analysis, secondary analysis and subcellular predication of MdPAO |
利用在线网址WoLF PSORT预测MdPAO亚细胞定位, 呈现出明显的亚家族特征, 且分布广泛(表3)。亚家族Ⅰ 中MdPAO3位于内质网; 亚家族Ⅲ 中的MdPAO1~2位于内质网, 其中 MdPAO7位于叶绿体; 亚家族Ⅳ 中MdPAO4、6和8位于过氧化物酶体, 而MdPAO5位于叶绿体。
利用在线网站GSDS和MEME对MdPAO的基因结构和保守基序进行了分析, 如图4所示。结果发现, 同一亚家族的成员表现出相似的基因结构和蛋白保守基序分布, 比如亚家族Ⅲ 中MdPAO1~2和MdPAO7都只含有1个外显子, 无内含子和5'/3'非翻译区, 都拥有motif1、motif3、motif4、motif6、motif7、motif8、motif9和motif10; 亚家族Ⅳ 中MdPAO4~6和MdPAO8拥有10个或13个外显子, 都拥有motif1、motif2、motif4和motif9, 但也存在差异, 其中MdPAO5缺失motif6, MdPAO4和MdPAO8缺失motif5。另外, 亚家族Ⅰ 中的MdPAO3具有10个外显子, 拥有motif1和motif6。
利用PLANTCARE在线网址分析启动子及其上游序列的顺式作用元件, 预测基因的功能, 结果如图5所示, 8个PAO都具有与光响应相关的顺式作用元件; 其次, 茉莉酸甲酯响应元件(methyl jasmonate, MEJA)除MdPAO5和MdPAO8外, 在其他成员中均有发现; 厌氧响应元件(anaerobic)除MdPAO5和MdPAO7外, 在其他成员中也均有发现; 另外, 在8个PAO中还存在与水杨酸(salicylic acid)、脱落酸(abscisic acid)、赤霉素(gibberellin)、生长素(auxin)、干旱(drought)和低温(low-temperature)相关的顺式作用元件。这些存在的调控元件说明MdPAO基因家族受到胁迫和多种激素的影响。
为了探讨MdPAO在不同组织中的表达特性, 试验采用GEO在线网站分析和实时荧光定量PCR两种方法。从GEO数据库中下载10个基因型(M69、M74、M20、M14、M49、GD、X8877、X4442、X4102和X3069)苹果的不同器官的基因表达量, 利用HemI软件制作热图。从图6中可以发现, MdPAO4在不同品种和器官中的表达量均显著高于其他家族成员, 其中在M49的叶和M74的花和果中表达量最高; MdPAO1~3和MdPAO7表达水平在M20的果中表达量最低; MdPAO1~2、MdPAO5~6和MdPAO8在不同杂交种的种子中表达量也很低。这些结果表明MdPAO在苹果不同品种和器官中的表达水平普遍存在, 为MdPAO基因的功能研究提供指导意义。
利用实时荧光定量PCR方法, 研究MdPAO基因在苹果长富2号不同组织器官中(茎、花、果、叶)的表达模式, 以探究MdPAO基因在苹果生长发育过程中的作用。由于MdPAO基因结构保守, 因此8个基因共设计出6对引物, 如表1所示[45]。MdPAO组织特异性结果表明(图7), 同一亚家族的MdPAO1/2和MdPAO7在4个不同组织中的表达模式相似, 在茎和果中的转录水平显著高于花和叶中的; 同一亚家族的MdPAO5/6和MdPAO8在4个不同组织中的表达模式也相似, 在花中表达量显著高于其余3个组织器官; MdPAO3在花和叶中的表达量显著高于茎中的, 在果中的表达量也较高; MdPAO4在花中的表达量显著高于茎和叶中的, 在茎中的表达量也较高。
为了研究苹果MdPAO在PA代谢中的潜在作用, 通过外源Spd处理, 在植物生理分化3个时期检测MdPAO的表达量。结果如图8所示, 所有MdPAO都响应Spd处理, 并具有相似的表达模式。进入生理分化期后, MdPAO的转录水平均是先上升后下降, 并在生理分化中期50 d达到最大值。除了MdPAO4以外, 其余MdPAO的表达水平在生理分化期间均有不同程度的上调, 其中MdPAO1/2在50和70 d显著高于对照, MdPAO4、MdPAO7和MdPAO8在30和50 d显著高于对照, 而MdPAO3通过外源Spd处理后, 没有显著变化。
大量的研究已经表明, PAO在植物生长和发育中起到至关重要的作用。拟南芥等模式植物对PAO家族成员的鉴定和系统进化的机制研究已经比较清楚, 在苹果中前人也有报道[16], 但关于苹果PAO基因家族的鉴定、特征和功能的系统性分析还未见报道。本实验共鉴定出8个苹果PAO家族成员, 并系统分析了其进化关系、理化性质、基因结构、启动子顺式作用元件和组织表达特异性等。
本研究从苹果基因组中共鉴定出8个MdPAO基因, 高于拟南芥、水稻、甜橙和番茄中的成员数量[6, 7, 12, 14], 这可能与苹果基因组较大和全基因组复制有关[46]。本实验对8个MdPAO蛋白的理化性质进行了分析, 发现均具有较好的稳定性且呈酸性, 大部分MdPAO蛋白是亲水蛋白。系统进化树分析显示, MdPAO与拟南芥和番茄等双子叶植物的PAO蛋白亲缘关系更近, 可能来源于同一祖先。植物中的PAO通常分为4个亚家族[7, 16, 17], 本实验中系统进化树分析结果表明, 8个MdPAO分为3个亚家族, 而没有属于亚家族Ⅱ 的成员。多重蛋白序列比对、蛋白结构、亚细胞定位预测和基因结构分析均显示出同一亚家族的成员具有相似的规律特征, 不同亚家族的成员之间呈现多样性, 这可能与MdPAO的进化密切相关。另外, 8个MdPAO基因启动子上富含茉莉酸甲酯、水杨酸、脱落酸、赤霉素和生长素等激素响应元件, 并拥有响应逆境胁迫相关的顺式作用元件, 暗示MdPAO可能参与涉及植物激素和逆境胁迫密切相关的多个生理过程。
通过GEO数据库和实时荧光定量PCR分析了MdPAO成员在不同器官和组织中的表达模式, GEO数据库的数据表明, MdPAO成员在苹果不同品种和器官中普遍表达, 并具有差异性。本研究利用实时荧光定量PCR探究了MdPAO成员在不同组织中的表达情况, 发现8个MdPAO在茎、花、果和叶均有表达。MdPAO3、MdPAO4、MdPAO5/6和MdPAO8在花中有较高的表达水平, 推测它们在花的生长发育过程中起到重要作用; MdPAO1/2和MdPAO7在茎和果中有较高的表达水平, 推测它们发挥功能的部位可能在茎和果中。进一步探索MdPAO基因如何在各组织中发挥功能, 还需要进行更系统的分子生物学实验研究。
基于Spd处理后MdPAO的表达数据表明, MdPAO在PA代谢途径中发挥着重要作用。根据MdPAO成员所在亚家族特征, 外源Spd处理后, MdPAO可能参与PA的反向转化途径, 促进Spm向Spd、Spd向Put的逆转化, 从而维持PA的稳态。MdPAO3没有显著变化, 推测其不响应外源Spd处理, 但有待进一步验证。MdPAO在PA代谢中的确切作用还需要使用转基因技术或使用蛋白质和相关底物的生化反应来阐明。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|








